DIAGNÓSTICO LABORATORIAL DE INFECCIONES POR CHIKV
Anuncio

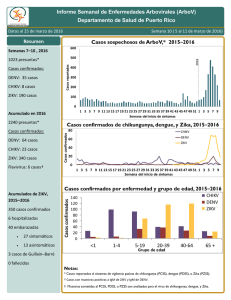
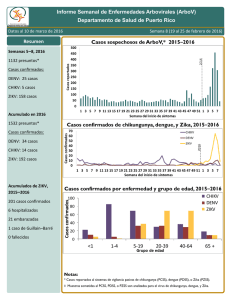
VIGILANCIA DEL VIRUS CHIKUNGUNYA POR LABORATORIO EN ARGENTINA Bioq. Victoria Luppo Laboratorio de Arbovirus Instituto Nacional de Enfermedades Virales Humanas (INEVH) “Dr. J.I.Maiztegui”- ANLIS, Pergamino-Argentina CHIKUNGUNYA Virus del género Alphavirus y Flia Togaviridae Deriva de Makonde: “aquel que se encorva” Son esféricos, con una nucleocápside dentro de una envoltura lipoproteica. Son virus con genoma de RNA de polaridad positiva de aprox. 11.5 KB Genes proteínas no estructurales: replicación del virus; genes de proteínas estructurales: implicadas en el reconocimiento con el sistema inmune (epítopes específicos) Proteínas no estructurales Proteínas estructurales Está Relacionado Genéticamente Y Serológicamente a O’nyong-nyong, Igbo Ora Y En Menor Medida Mayaro Y Ross River • 1 Serotipo •3 Genotipos Principales: •Africa Occidental (selvatico) •Africa Central/Este/Sur (ECSA) •Asiatico-Americano Selección, recolección y envío de muestras Selección de muestras a procesar: Muestras de pacientes con inicio agudo de fiebre >38.5º C, artralgia grave o artritis no explicada por otra condición médica, que reside o haya visitado áreas con circulación de CHIKV. Tipo de muestra: Suero de Fase aguda: hasta 8 días tras el inicio de síntomas Suero de Fase convalesciente: 10-15 días tras el inicio de síntomas LCR: casos de meningoencefalitis. Conservación de la muestra: -Refrigerada si se procesa dentro de las 48 hs -Congelada si se procesa después de las 48 hs Envío de la muestra -Con hielo seco (en lo posible), o con geles refrigerantes para asegurar la cadena de frío -Siempre con la ficha clínico-epidemiológica completa (fecha de inicio de los síntomas, Fecha toma de muestra, Historia de viajes, antecedentes de vacunación, etc) -Triple envase VIREMIA Y CINETICA DE ANTICUERPOS EN INFECCIONES POR CHIKV DIAS DE EVOLUCIÓN Técnicas de laboratorio para CHIK Aislamiento viral e identificación por IF o RT-PCR Detección de genoma viral (RT-PCR convencional y en Tiempo Real ) Detección de anticuerpos contra proteínas virales: Elisa IgM/IgG, Neutralización por reducción del número de placas. Inactivación 1er paso extracción del RNA (BSL2) Algoritmo para detección de CHIKV, DENV y requerimientos de Bioseguridad Muestra de Caso sospechoso de CHIKV y DENV Serología en casos RT-PCR (-) CHIK en BSL2 < 8 días de evolución ELISA IgM DENV (desde 4 dpi) RT- PCR DENV y CHIKV Positivo Negativo Positivo Negativo IgM CHIKV Secuenciación – Filogenia Positivo Aislamiento Viral (BSL3) Estudiar PAR SEROLÓGICO por Neutralización r técnica de para CHIK y otros Alphavirus (BSL3) Negativo Solicita Segunda muestra para repetir serología Estudiar por técnica de neutralización para DEN y otros Flavivirus (BSL2, virus quiméricos para SLEV y WN, cepa vacunal YFV) Algoritmo para detección de CHIKV, DENV y requerimientos de Bioseguridad Caso sospechos de DENV y CHIKV Serología en BSL2 ≥ 8 días de evolución ELISA IgM DENV y CHIK V Positivo CHIKV Estudiar PAR SEROLÓGICO por técnica de neutralización para CHIK y otros Alphavirus ( BSL3) *Estudio de segunda muestra para descartar posible infección secundaria de DENV sin IgM (Clínica y Epidemiología) Positivo DENV Negativo DENV y CHIKV Estudiar PAR SEROLÓGICO por técnica de neutralización para DENV y otros Flavivirus Se descarta caso de DENV* o CHIKV (BSL2, virus quiméricos para SLEV y WN, cepa vacunal YFV) •Búsqueda de otros Arbovirus TÉCNICAS DIRECTAS AISLAMIENTO VIRAL Gold Stándard de los métodos directos Muestra indicada: suero de fase aguda (< de 8 días de evolución y perfectamente conservada) Inoculación en lineas celulares suceptibles: BHK-21, HeLa, Vero y C6/36. Efecto citopático al 3er día. Confirmación por técnica de IF , RT-PCR convencional o en real time del sobrenadante celular. Aislamiento en laboratorio BSL-3 de cepas obtenidas en el INEVH de casos importados Aislamiento de cepas de CHIKV en el INEVH (BSL 3) 1. 2. 3. 4. Muestras de Suero de casos sospechosos (< 8días de evolución) Se aislaron en celulas Vero Identificación viral por RT-PCR Real Time CHIKV Titulación por UFP Secuenciación de un fragmento de Genoma Viral y filogenia viral Se han obtenido stocks virales con titulos ˜ 10 7 ufp /ml: • cepa Asiatica (aislamiento viral de suero de un viajero detectado en Argentina) Celulas Vero C 76 RT-PCR en tiempo real CHIKV Inicialmente se usó un protocolo diseñado por el Dr. Lanciotti 2007 (sensibilidad: 1UFP o 50 copias genómicas) Diseño de sonda y primer en base a la cepa prototipo S27 Detecta todos los genotipos West Africa, ECSA y Asia No hay reactividad para otros Alphavirus (ONN, Ross River, Mayaro, EEE, WEE, VEE, Semiliki Forest, Sinbis). RT-PCR en tiempo real CHIKV • Recientemente se han diseñado 2 sets de primers y sonda más específicos para el genotipo asiático circulante (Fort Collins- CDC) (sin publicar aún) • Se analizaron en el INEVH diluciones seriadas de una de las cepas aisladas en el INEVH procedente de República Dominicana Los sets 2 y 3 tienen mayor sensibilidad que el set 1, por lo cual se recomienda para el análisis de las cepas que circulan actualmente en América AAAAAAAAAAA nsP1 nsP2 nsP3 nsP4 C E2 E1 No estructural estructural Sets de primer y sondas • Set 1 (ECSA) 6856F 6981c 6919-FAM • Set 2 (Asiático-América) 3855F 3957c1,2 3996-FAM1,2 • Set 3 (Asiático-América) 856F 962c 908-FAM RT-PCR convencional genérica para Alphavirus Oligonucleótidos degenerados en la región nsp4 (195pb) Detección de EEE, WEE, VEE, CHIK, Ross River, Sinbis, Semiliki Forest Para identificar el Alphavirus se requiere la secuenciación genómica AAAAAAA nsP1 nsP2 nsP3 nsP4 No estructural C E3 E2 E1 estructural Caracterización Filogenética de cepas circulantes CHIKV detectado en Argentina- INEVH 2014 Identificación del Genotipo Asiático en viajeros procedentes del Caribe Análisis bayesiano, 1200 nt en la región E ; 155 pb región nsp4. Árboles de topología similar Congreso Argentino de Virología CAV,2015 TÉCNICAS SEROLÓGICAS ELISA de Captura de Ac IgM Desde el 2011 el INEVH cuenta con la técnica de Elisa IgM de captura puesta a punto y evaluada externamente a través de proficiencia del CDC Permite detectar infección actual o reciente Presenta cruce serológico con otros alfavirus del mismo grupo ej: virus Mayaro, Ross River, Onyong nyong) Metodología in house que utiliza reactivo producido en INEVH ELISA de captura IgM CHIK (P/N) CHIK RR ONN VEE MAY EEE 12.3 1.5 15.3 0.89 1.9 1.2 10.6 1.2 13.9 1.1 1.9 1.3 14.5 1.7 17.3 1.1 3.1 0.89 20.4 0.92 20.7 1.5 7.1 1.8 26.6 4.9 27.4 2.2 1.7 1.5 21.2 1.5 24.7 2.2 1.2 1.6 15.3 1.5 8 1.6 1.9 2.2 31.3 1.5 24.6 1.7 2.9 1.8 22 1.6 15.9 1.8 1.4 1.4 18.8 0.59 13.1 1.2 1.5 1.1 34.3 3.7 22.6 1.6 2.8 9.1 Fuente: CDC IgM Capture ELISA 1. Anti IgM humano (4°C toda la noche) 2. Agregar suero de paciente en la dilución adecuada (37°C , 1 Hora) 3. Agregar Antígeno (chik y normal para la misma muestra) ( 4°C toda la noche) 4. Agregar el Ac monoclonal conjugado con peroxidasa rábano (37°, 1 Hora) 5. Adición del Sustrato TMB. Dejar 30 min a 37°C y solución de Stop. Leer a 450 nm Fuente: CDC Inoculación viral Linea Celular C6 / /BHK-21 36 VeroC76 Producción de antígenos: “Slurry” para ELISA IgM Incubación x días, 34ºC Monitoreo por ECP y/o IFI 100 % Infección Congelamiento -70ºC Descongelamiento Sonicado y centrifgado ANTIGENO p/ ELISA IgM Inactivación por irradiación gamma Puesta a punto de la PRNT para confirmación serológica de CHIKV Técnica serológica de referencia para confirmar el diagnóstico Aumento en 4 veces el título entre muestra aguda/convalesciente. Estudiar especificidad con panel de Alphavirus Se tituló stock viral de MAY para incorporar al panel INCORPORACIÓN DEL DIAGNÓSTICO DE CHIKV EN LA RED NACIONAL DE LABORATORIOS PARA DIAGNOSTICO DE DENGUE Y OTROS ARBOVIRUS INEVH Confirmación, Control de calidad, trasferencia de tecnología y capacitación Puesta a punto de PRNT, producción de antígenos para ELISA, producción de controles positivos para PCR Incorporación del Evento CHIK en SIVILA Puesta a Punto de Real Time en laboratorios de la Red Puesta a Punto de RTPCR Genérica de Alfavirus en laboratorios sin equipos Real Time ¡MUCHAS GRACIAS POR SU ATENCIÓN! Bioq. Victoria Luppo [email protected] Dra Alejandra Morales [email protected] Lic Cintia Fabbri [email protected]