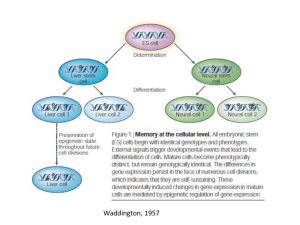
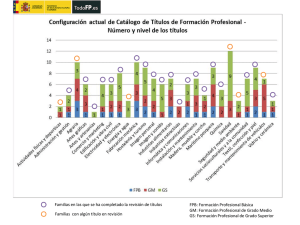
Caracterización y análisis funcional de las histonas H2A y H2B de
Anuncio

!"#!$" Caracterización y análisis funcional de las histonas H2A y H2B de Toxoplasma gondii % % & ' () & &*$) 2009 Agradecimientos !"#$%# # & ' " ( ) * +,+ # & -) & ( .- #&/$ 0'&' # 123 4 ! 5 678 # & 9 / # ! 5+ # % /" # # 5 $+ : +&#/ ; < = 6 5 6 57 5 # /8 5 ( ! >#$ "/ ( # 5 = (!5 66 ii # %40 $> 5 ! ) 5 # %%2?%40 $> & $ # )5 ( #$'4%$ 0#4$=0 (# $'? $'4%$ 0?4) 8= ( @#4$=0;%$0AB?CDDEB<14#/4%>?4%#%&;E"AE#%AFCEG3?AE< iii Indice Agradecimientos ....................................................................................................................... ii Indice............................................................................................................................................ 1 Publicaciones ............................................................................................................................. 4 Abreviaturas y palabras tomadas del inglés ....................................................................... 5 Resumen ...................................................................................................................................... 7 Capítulo 1: 1. Toxoplasma gondii .............................................................................................................. 10 1.1. Caracterísitcas generales ....................................................................................... 10 1.2. Genotipos existentes .............................................................................................. 10 1.3. Ciclo de vida del parásito ........................................................................................ 11 1.4. Interconversión taquizoito-bradizoito ....................................................................... 13 1.5. Regulación de la transcripción ................................................................................ 17 2. HISTONAS ............................................................................................................................... 18 2.1. Características generales ....................................................................................... 18 2.2. El lenguaje de las modificaciones covalentes de las histonas ................................. 19 2.2. Variantes y canónicas ............................................................................................. 21 2.4. Variantes de H2A .................................................................................................... 22 3. Lenguaje de las histonas en Toxoplasma....................................................................... 25 3.1. Evidencias de la presencia de mecanismos epigenéticos ....................................... 25 OBJETIVOS: ................................................................................................................................ 29 General: ......................................................................................................................... 29 Específicos: ................................................................................................................... 29 Capítulo 2: !" 2. 1. Introducción ..................................................................................................................... 31 2. 2. Resultados ......................................................................................................................... 33 2. 2. 1. Identificación y clonado ...................................................................................... 33 2. 2. 2. Análisis filogenético ........................................................................................... 36 2. 2. 3. Antigenicidad de H2Bv en pacientes con toxoplasmosis .................................... 37 2. 2. 4. Obtención de anticuerpos específicos................................................................ 37 2. 2. 5. Inmunodetección de las H2Bs............................................................................ 38 2. 2. 6. Análisis de histonas purificadas ......................................................................... 40 1 2. 2. 7. Expresión de las histonas H2Bs de T. gondii ..................................................... 41 2. 3. Discusión ............................................................................................................................ 44 Capítulo 3: #$ #%& 3.1. Introducción ...................................................................................................................... 47 3.2. Resultados .......................................................................................................................... 49 3. 2. 1. Identificación, clonado y análisis de secuencia .................................................. 49 3. 2. 2. Obtención de anticuerpos específicos................................................................ 53 3. 2. 3. Inmunodetección de las histonas de la familia H2A ........................................... 55 3. 2. 4. Perfiles de expresión de las histonas de la familia H2A ..................................... 57 3. 2. 5. H2AX y daño del ADN en taquizoitos ................................................................. 59 3.2.6. La sobreexpresión de H2AX y su papel en la replicación del parásito.................. 62 3.1. Discusión ............................................................................................................................. 64 Capítulo 4: ' &( 4.1. Introducción ...................................................................................................................... 69 4.2. Resultados .......................................................................................................................... 71 4.2.1 Interacciones entre las histonas H2As y H2Bs ...................................................... 71 4.2.2 H2As y H2Bv: acetilación y asociación con histonas acetiladas ............................ 73 4.2.2 Asociación diferencial de H2A y H2B variantes a genes activos e inactivos.......... 75 4.2.4 H2AX se encuentra asociada a TgIRE, un elemento repetitivo al extremo de los cromosomas .................................................................................................................. 75 4.3 Discusión .............................................................................................................................. 78 Capítulo 5: )( Capítulo 6: *(& 6.1. Cultivo de parásitos ................................................................................................ 87 6.2. Análisis de secuencia ............................................................................................. 88 6.3. Análisis de Neighbor-joining.................................................................................... 88 6.4. Amplificación y clonado de las diferentes histonas.................................................. 89 6.5. Análisis de expresión .............................................................................................. 90 6.6. Expresión y purificación de las histonas recombinantes.......................................... 92 6.7. Producción de anticuerpos...................................................................................... 93 6.8. Purificación de histonas .......................................................................................... 93 6.9. Análisis de histonas purificadas por TAU-PAGE y MALDI-TOF-TOF. ..................... 94 2 6.10. Análisis de Western Blot ....................................................................................... 94 6.11. Inmunofluorescencia indirecta (IFI) ....................................................................... 96 6.12. Determinación de la especificidad de los anticuerpos ........................................... 97 6.13. Coinmunoprecipitación.......................................................................................... 98 6.14. Inmunoprecipitación de cromatina (ChIP) ............................................................. 99 6.15. Tratamiento con peróxido de hidrógeno .............................................................. 101 6.16. Construcción de plásmidos de expresión en Toxoplasma ................................... 102 6.17. Transfección de parásitos ................................................................................... 102 6.18. Ensayo de replicación. ........................................................................................ 103 Bibliografía............................................................................................................................... 104 3 +, Publicaciones Parte de los resultados de esta tesis han sido presentados en: Publicaciones “Toxoplasma H2A Variants Reveal Novel Insights into Nucleosome Composition and Functions for this Histone Family”. Maria C. Dalmasso, David O. Onyango, Arunasalam Naguleswaran, William J. Sullivan Jr. and Sergio O. Angel. Journal of Molecular Biology (2009) 392(1):33-47. “Toxoplasma gondii has two lineages of histones 2b (H2B) with different expression profiles”. María C. Dalmasso, Pablo C. Echeverria, María P. Zappia, Ulf Hellman, Jean François Dubremetz and Sergio O. Angel. Mol Biochem Parasitol (2006) 148(1):103. Presentaciones a congresos “Toxoplasma gondii histones H2A: novel features about H2AX”. Dalmasso MC, Sullivan WJ and Angel SO. XXIII Reunión Annual de la Sociedad Argentina de Protozoología -SAP- Rosario, Argentina (Noviembre, 2008). “Análisis de las interacciones y modificaciones postraduccionales de las histonas H2A y H2B de Toxoplasma gondii”. Dalmasso MC, Sullivan WJ and Angel SO. XXII Reunión Annual de la Sociedad Argentina de Protozoología -SAPChascomús, Argentina (Noviembre, 2007). “Toxoplasma gondii histone H2Bv interacts with H2A.Z but not with H2A.X”. Dalmasso MC and Angel SO. Ninth International Congress of Toxoplasmosis. Montana USA (Junio-Julio, 2007). “Caracterización de las histones H2A de Toxoplasma gondii”. Dalmasso MC, Echeverria P and Angel SO. XXI reunión anual de la Sociedad Argentina de Protozoología. Huerta grande, Córdoba (Octubre 2006). “Cloning and caracterization of Toxoplasma gondii histones 2A”. Dalmasso MC, Echeverria P, Hellman U, Dubremetz JF and Angel SO. Eighth International Congress on Toxoplasmosis. Corsica - France (Mayo 2005). 4 , Abreviaturas y palabras tomadas del inglés aa.: aminoácidos AN: número de acceso del gen en el GeneBank. Del inglés “Accession Number”. AcH3: histona H3 acetilada. AcLys: lisina acetilada. ADN: ácido desoxiribonucleico. ADNc: ácido desoxiribonucleico copia del ARN. ADNg: ácido desoxiribonucleico genómico. ARN: ácido ribonucleico. ATM: kinasa ataxia telangiectasia mutada. ATR: kinasa ATM relacionada a Rad3. BAG1: antígeno de bradizoito. Del inglés “Bradyzoite Antigen 1”. BSA: albúmina sérica bovina. Del inglés “ Bovine Serum Albumin” CDS: secuencia codificante. Del inglés “ CoDing Sequence”. CT: número de ciclo de la PCR en el cual la cantidad de producto amplificado cruza el umbral establecido. Del inglés “ Cycle Threshold”. ChIP: inmunoprecipitación de cromatina. Del inglés: “Chromatin Immuno Precipitation”. DBL: lectina de Dolichos biflorus. Del inglés “Dolichos biflorus lectin”. Tiene la capacidad de unirse a la pared del quiste. DNA-PK: ADN protein kinasa. dNTP: deoxi nucleótidos trifosfatos. DAPI: 4,6-diamidino 2-fenil indol. Marcador de núcleo (fluorescente) DSB: daño en el ADN generado por quiebre de las dos hebras. Del inglés “Double Strand Brakes”. ELISA: Ensayo por inmunoabsorción ligado a enzimas. Del inglés "Enzyme-Linked ImmunoSorbent Assay". EST: marcador de secuencia expresada. Del inglés “Expressed Sequence Tag”. FTG: Factores de Transcripción Generales. GST: glutatión tio-transferasa. γH2AX: histona H2AX fosforilada. HAT: Histona Acetil Transferasa. HDAC: Histona Desacetilasa. H4K20me1: Histona H4 monometilada en la lisina 20. 5 , hxgprt: hipoxantina xantina guanina fosforibosil transferasa. Del inglés “hypoxanthinexanthine-guanine phosphoribosyl transferase”. IFI: Inmunofluorescencia Indirecta. IP: inmunoprecipitación. IPTG: Isopropil ȕ-D-1-tiogalactopiranósido. KO: mutante nula. Del inglés “knock out”. kDa: kilodalton LB: Medio de cultivo para bacterias Luria-Bertoni. LDH2: Lactato Deshidrogenasa 2. Este gen es específico de bradyzoitos. OD(λλ=600): densidad óptica medida a una longitud de onda de 600 nm. Del inglés “optic density”. oligo(dT)12-18: oligonucleótido compuesto por 12 a 18 timidinas. o.n.: toda la noche. Del inglés “ over night”. pb: pares de bases. PBS: solución tampón de fosfato salino. Del inglés “ Phosphate Buffer Saline”. PCR: reacción en cadena de la polimerasa. Del inglés “ Polimerase Chain Reaction”. pI: punto Isoeléctrico. qPCR: análisis de cuantificación absoluta por PCR en tiempo real. Del inglés “quantitative PCR”. qRT-PCR: análisis comparativo por PCR en tiempo real. Del inglés “quantitative retrotranscription - PCR”. MMS: metano sulfonato de metilo. Del inglés “Methyl Methane Sulfonate”. MNasa: endocucleasa micrococcal. RT-PCR: transcripción reversa del RNA seguida de reacción en cadena de la polimerasa. Del inglés “ retro-transcription – PCR”. SAG 1: antígeno de superficie. Del inglés “ Surface Antigen 1”. Es un gen específico de taquizoito. SDS-PAGE: electroforesis en gel de poliacrilamida conteniendo dodecil sulfato sódico. Del inglés “Sodium Dodecyl Sulfate – Poly Acrylamide Gel Electrophoresis”. SNP: Polimorfismo de un solo nucleótido. Del inglés “Single Nucleotid Polimorfism”. TAU-PAGE: Electroforesis en gel de poliacrilamida conteniendo Triton, ácido acético y urea. Del inglés “ Tritón-Acetic-Urea – PAGE”. TBS: solución tampón Tris-salino. Del inglés “ Tris Buffer Saline”. TEMED: N,N,N’,N’ tetrametil etilen diamina. TSS: sitio de inicio de la transcripción. Del inglés “Transcriptional Start Site”. WT: cepa salvaje. Del inglés: “ Wild Type”. 6 - Resumen Toxoplasma gondii posee la capacidad de diferenciarse entre la forma de replicación rápida (el taquizoito), y la de replicación lenta o nula (el bradizoito), que forma quistes. Esta interconversión regula que se establezca una infección crónica y la reaparición de la enfermedad. Si bien este proceso se encuentra regulado a nivel transcripcional, no se conocen los mecanismos que regulan la transcripción en el parásito. Varios trabajos han demostrado que los mecanismos epigenéticos, como la acetilación y metilación de histonas H3 y H4, se encuentran conservados en T. gondii. Nuestra hipótesis de trabajo fue que las histonas H2A y H2B estarían involucradas en la regulación de la transcripción y diferenciación del parásito. Con el objetivo de esclarecer esta idea, se identificaron y caracterizaron los miembros de las histonas H2A y H2B de T. gondii. Se detectaron tres H2B en el genoma de T. gondii: H2Bv, H2Ba y H2Bb. Un análisis de Neighbor-Joining, incluyendo las secuencias de H2B de otros Apicomplexas, mostró que éstas se agrupan en dos clados diferentes, uno que contiene a la H2Bv y el otro a H2Ba y H2Bb. A diferencia de otros organismos todos los parásitos Apicomplexas analizados presentan una H2B variante. Por ensayos de RT-PCR semicuantitativa, se observó que sólo se expresan h2bv y h2ba, siendo h2bv la mayoritaria. A su vez, los niveles de transcripto de H2Ba disminuyen en bradizoitos, mientras que los de H2Bv son iguales en taquizoitos y bradizoitos; confirmando la calidad de histonas canónica y variante, respectivamente. Se desarrolló un anticuerpo específico para H2Bv. Los ensayos de Western blot e Inmunofluorescencia indirecta mostraron que los niveles de proteína de esta histona en taquizoitos y/o bradizoitos son iguales y que en ambos estadios se encuentra en el núcleo. Un estudio de histonas purificadas por MALDI-TOF y microsecuenciación, reveló la presencia de las histonas H2A, H3, H4 y H2Bv en las bandas analizadas, mientras que H2Ba no pudo ser identificada. Estos resultados estarían confirmando que la histona variante es la H2B mayoritaria. Por otro lado, se encontraron tres H2A en el genoma de T. gondii: H2A1, H2AX y H2AZ. Se obtuvieron anticuerpos específicos para cada una. Estos se utilizaron en ensayos de Western blot de histonas purificadas, revelando que las tres H2As se encuentran presentes en taquizoitos y que H2AZ es la minoritaria. El estudio de los niveles de transcripto en taquizoitos y bradizoitos in vitro por RT-PCR en tiempo real (qRT-PCR) mostró un aumento de h2a1 y h2aX en bradizoitos, mientras que h2aZ permanece constante. Sin embargo, cuando se analizan en bradizoitos in vivo, solo se observa la 7 - expresión de h2aX y h2aZ. Como la mayoría de los bradizoitos in vitro se encuentran arrestados en S tardía-G2/M, mientras que los in vivo en G0, podemos concluir que H2A1 es una histona canónica. Por otro lado, h2aZ y h2aX mantienen los mismos perfiles de expresión en ambos tipos de bradizoitos, confirmando su calidad de variantes. A través de la utilización de un anticuerpo comercial que reconoce la H2AX fosforilada (γH2AX), fue posible determinar que la H2AX de T. gondii se encuentra fosforilada en taquizoitos y que los niveles de γH2AX aumentan en parásitos tratados con H2O2. Además, se observó por qRT-PCR que este daño oxidativo estimula la transcripción de h2a1 y h2aX. Con el fin de obtener mayor información a cerca de la función de H2AX, se obtuvieron parásitos que sobreexpresan esta histona variante. Los experimentos preliminares indicaron que H2AX estaría involucrada en la replicación del parásito. Se analizó la composición de los nucleosomas mediante ensayos de coinmunoprecipitación (coIP) con los anticuerpos específicos para H2AX, H2AZ y H2Bv. En taquizoitos H2Bv se encuentra formando dímeros con H2AZ pero no con H2AX. A su vez, H2AZ y H2AX no se encuentran en el mismo nucleosoma. Por lo tanto, H2Bv-H2AZ y H2AX se encuentran en nucleosomas diferentes. Finalmente, se analizó la asociación de las variantes H2AX, H2AZ y H2Bv con histonas acetiladas y su localización genómica por ensayos de coIP e inmunoprecipitación de cromatina (ChIP). Las variantes H2AZ y H2Bv están acetiladas y/o asociadas a otras histonas acetiladas y se encuentran enriquecidas en regiones transcripcionalmente activas. Por el contrario, H2AX se asocia muy levemente con histonas acetiladas y se encuentra enriquecida en regiones promotoras inactivas o silentes. Todos estos resultados fuertemente sugieren que las histonas H2A y H2B variantes forman parte de diferentes mecanismos epigenéticos que se encontrarían regulando diversos procesos celulares en T. gondii. 8 Capítulo 1 )#$. 1. Toxoplasma gondii 1.1. Características generales Toxoplasma gondii es uno de los parásitos mayormente distribuidos, tanto a nivel de variedad de hospedador como de distribución geográfica. Es capaz de infectar a casi todos los animales homeotermos. Este parásito pertenece al phylum Apicomplexa, el cual es un amplio grupo de protistas intracelulares. La mayoría de ellos presentan una organela particular denominada apicoplasto y un complejo apical involucrado en la invasión a las células hospedadoras. Este grupo presenta una gran diversidad, incluyendo patógenos de humanos como ser los pertenecientes al género Plasmodium (causante de la malaria) y Cryptosporidium, así como también patógenos de animales, entre ellos los pertenecientes al género Eimeria y Sarcocystis. La población infectada en el Reino Unido y Estados Unidos de Norteamérica se estima entre 16-40%; mientras que en América del Sur, Central y Europa entre 59-80%; por lo que presenta gran importancia socio-económica (Hill et al. 2005). En Argentina, la prevalencia de anticuerpos es elevada, aunque habría diferencias a nivel geográfico (Ej.: 23.8% en Chaco y 51.7% en Buenos Aires) (Durlach et al. 2008). Es interesante destacar que dada la facilidad que presenta T. gondii para ser cultivado in vitro y para ser estudiado mediante el uso de técnicas de genética molecular, este parásito se ha convertido en los últimos tiempos en un organismo modelo para el estudio dentro del phylum Apicomplexa. 1.2. Genotipos existentes Toxoplasma presenta una población clonal compuesta por tres linajes, designados Tipo I, Tipo II y Tipo III. Estos linajes clonales emergieron de eventos de recombinación meiótica de un ancestro común (Grigg and Suzuki 2003). Se han descripto varias características biológicas relacionadas con el linaje. La más reconocida es la virulencia en ratones. Las cepas de Tipo I son virulentas, provocan una diseminación generalizada del parásito y la muerte de los ratones en menos de 10 días luego de la inoculación con una dosis menor a 10 taquizoitos. En cambio las de Tipo II y III son avirulentas. Los ratones sobreviven a la infección con cepas de Tipo II (Dosis letal del 50% (DL50) > 103 taquizoitos) y la diseminación del parásitos es mucho menos extensiva. Mientras que, si 10 )#$. bien se considera a las cepas de Tipo III como avirulentas, pueden provocar un deterioro progresivo y muerte de los ratones, semanas o meses después de la inoculación. Cabe destacar que la virulencia varía entre los distintos hospedadores intermediarios, por ejemplo: los parásitos Tipo I no son patogénicos en ratas (Saeij et al. 2005). En cultivos in vitro las cepas Tipo I crecen más rápido que las Tipo II y III y presentan una tasa de interconversión de taquizoitos a bradizoitos mucho menor. La mayor tasa de crecimiento de los parásitos Tipo I se debe a un aumento de la invasión más que a un acortamiento del tiempo de replicación (Weiss and Kim 2007). 1.3. Ciclo de vida del parásito T. gondii posee un ciclo de vida complejo (Figura 1.1.2). Presenta un ciclo sexual que se desarrolla únicamente en el intestino del gato, el cual es el hospedador definitivo y un ciclo asexual que puede ocurrir en cualquier hospedador intermediario: mamíferos (incluyendo a los felinos) y aves. Estos dos ciclos teóricamente pueden coexistir independientemente (Boothroyd et al. 1998). Existen cuatro formas invasivas de T. gondii: taquizoito, bradizoito, merozoito y esporozoito. Los dos primeros están asociados con el hospedador intermediario y los últimos dos con el hospedador definitivo. Ciclo de vida en el hospedador definitivo El ciclo comienza cuando un miembro de la familia felina ingiere un ooquiste maduro del medio ambiente o tejidos con quistes tisulares. Las paredes de los mismos son disueltas por las enzimas digestivas liberando esporozoitos o bradizoitos, respectivamente, que infectan a las células epiteliales del intestino delgado. El ciclo de vida en el hospedador definitivo se encuentra limitado a estas células. En todos los estadios, los parásitos se encuentran dentro de una vacuola parasitófora ajustada de membrana muy fina. El desarrollo asexual se produce mediante un proceso denominado endopoligenia (Figura 1.1.1). Este involucra el crecimiento del parásito y repetidas divisiones nucleares, dando origen a una célula multinucleada llamada meronte o esquizonte. Luego, ocurre la división de las organelas y formación de tantas células hijas como núcleos presentes (de 4 a 20), denominadas merozoitos (Ferguson et al. 1974). Estos merozoitos son liberados al lumen del intestino donde pueden reinvadir otros enterocitos. Morfológicamente, los merozoitos son similares a los taquizoitos pero se distinguen en que pueden diferenciarse en gametas. Luego de un número desconocido de ciclos asexuales, ocurre el ciclo sexual del parásito, donde los merozoitos dan origen a microgametas (gameta masculina) y 11 )#$. macrogametas (gameta femenina). El merozoito invade un enterocito, se vuelve más esférico y pierde la mayoría de sus organelas apicales. Este estadio, trofozoito, a través de un proceso denominado microgametogonia forma múltiples (15-30) microgametas. A través del proceso de macrogametogonia, el trofozoito da origen a una macrogameta, la cual presenta la pared impermeable y resistente de los ooquistes. Se supone que la fusión de ambas gametas da origen a un cigoto, sin embargo ésto nunca se ha visualizado. Los ooquistes son liberados al medio ambiente en las heces del felino, donde ocurre la esporulación. Esta finalmente resulta en la formación de dos esporoquistes, cada uno con 4 esporozoitos haploides altamente infectivos, pudiendo sobrevivir en el ambiente por varios meses y posiblemente hasta años. Figura 1.1.1. Tipos de división en T. gondii. Endodiogenia, donde de un parásito se forman dos. Endopoligenia, donde se forman tantos parásitos como núcleos existan. Fuente: The Morrissett Lab (www.morrissettelab.bio.uci. edu). Ciclo de vida en el hospedador intermediario En los hospedadores intermediarios sólo ocurre el ciclo asexual, luego de la ingestión de ooquistes o tejidos con quistes. En este caso los parásitos invaden las células epiteliales y/o linfocitos y se diferencian en taquizoitos. Dentro de la vacuola parasitófora, los taquizoitos se replican rápidamente por un proceso denominado endodiogenia (Figura 1.1.1), el cual es similar a endopoligenia salvo que cada taquizoito da origen a dos taquizoitos. Esto ocurre repetidas veces hasta que se produce la lisis de la célula hospedadora. Los taquizoitos liberados al medio extracelular son capaces de infectar a cualquier célula nucleada que encuentren, replicarse y lisar a la célula hospedadora repitiendo el ciclo lítico. Este estadio coincide con la fase aguda de la toxoplasmosis o reactivaciones. Si la infección no es controlada por el sistema inmune, 12 )#$. los parásitos pueden dispersarse muy rápidamente hacia otros tejidos causando una toxoplasmosis generalizada cuyos efectos son siempre fatales (Frenkel 1967). La respuesta del sistema inmune frente a la infección provoca que los taquizoitos intracelulares se diferencien a bradizoitos, los cuales se replican de manera lenta o nula y forman una pared dando origen a los quistes titulares. De este modo permanecen invisibles ante el sistema inmune gracias a que la pared quistídica está compuesta principalmente por productos derivados de la célula huésped. Estos quistes aparecen a los 12-15 días postinfección y definen el estadio crónico de la toxoplasmosis (Weiss and Kim 2000). Estos se presentan como una importante fuente de infección para el próximo hospedador susceptible, al ser ingeridos mediante el consumo de carne cruda o mal cocida. Ooquistes No esporulados Ingestión Hospedador definitivo Felinos (esquizonte, merozoitos,macro y Quistes tisulares microgametas) (bradizoitos) Ooquistes Esporulados (esporozoitos) Contaminación de agua o alimentos Ingestión Hospedador intermediario Mamíferos y aves (taquizoitos) Ingestión Figura 1.1.2. Diagrama del ciclo de vida de T. gondii. Los componentes sexuales y asexuales del ciclo de vida son potencialmente independientes (Boothroyd, Hehl et al. 1998). En particular, la fase asexual puede teóricamente ciclar entre hospedadores intermediarios ad infinitum. 1.4. Interconversión taquizoito-bradizoito La infección toxoplásmica presenta una fase aguda generada por los taquizoitos y una fase crónica dada por la presencia de bradizoitos. El paso de una fase a la otra, determinada por el cambio de estadio del parásito, se debe a la presencia del sistema 13 )#$. inmune. Si éste falla, los bradizoitos pueden volver a convertirse en taquizoitos y de este modo reactivar la enfermedad. Por lo tanto, esta interconversión es crítica en la patogenia del parásito y el control de la infección. La infección con Toxoplasma en humanos puede resultar en encefalitis en pacientes inmunocomprometidos, coriorrenitis en pacientes inmuncompetentes o fetopatías y/o aborto a través de una transmisión congénita si la mujer embarazada es seronegativo al momento de la infección. Además, se ha estimado que en pacientes con VIH (Virus de Inmunodeficiencia Humana) sin tratamiento antirretroviral, el riesgo de manifestar síntomas por toxoplasmosis es mayor al 30% (Weiss and Kim 2007). La diferenciación de taquizoitos a bradizoitos requiere de la progresión en el ciclo celular, ya que ésta no ocurre si el crecimiento de los taquizoitos se encuentra bloqueado (Radke et al. 2001). El ciclo celular de los taquizoitos consiste en la fase G1 (con un contenido de ADN de 1N), la fase S (con un contenido de ADN de 1,8N), una mitosis [M] (con un contenido de ADN de 2N) que ocurre inmediatamente luego de la duplicación del ADN y finaliza con una citoquinesis [C] (Figura 1.1.3A y (Radke et al. 2001)). La población con un contenido de ADN de 2N es muy baja, indicando que no habría una fase G2. Esta característica del ciclo celular de taquizoitos cambia frente a la presencia de un estrés, cuando los ciclos de división del parásito se enlentecen y el desarrollo de bradizoitos se hace evidente. En estos parásitos en diferenciación aparece una nueva subpoblación con un contenido de ADN nuclear de 1,8-2N, que no sintetizan ADN activamente ni entran en mitosis; indicando la presencia de una fase S-tardía/G2 (Radke et al. 2003). La detección de marcadores de taquizoitos y bradizoitos en relación al contenido de ADN, reveló que el 85% de la población S-tardía/G2 presenta ambos marcadores. Esto sugiere que los parásitos en esta fase se encuentran en un estadio intermediario en la diferenciación a bradizoitos, ya que los parásitos extraídos de quistes tisulares in vivo se encuentran arrestados en G0 (toda la población contiene 1N de ADN) (Figura 1.3.1B). 14 )#$. Figura 1.1.3. Ciclo celular en taquizoitos y bradizoitos. A. Fases del ciclo celular en taquizoitos: G1 (crecimiento), S (síntesis), M (mitosis) y C (citoquinesis) y la representación gráfica de la morfología del parásito en cada una. Se observa el porcentaje de células en cada fase y el contenido de ADN: 1N en G1; 1,8N en S y 2N en M (Radke J.R. et al 2001). B. Modelo propuesto del ciclo celular en la diferenciación. En el pasaje a bradizoitos, aparece una fase S tardía-G2 con un contenido de ADN de 1.8N, donde se cree que se sintetiza lo necesario para la pared del quiste; mientras que los bradizoitos y esporozoitos maduros se encuentran arrestados en G0. Durante la recrudescencia, bradizoitos y esporozoitos primero se diferencian a taquizoitos (SAG1 +) y luego entran al ciclo celular (Radke J.R. et al 2003). Estudios in vitro revelaron que si los parásitos se cultivan en condiciones estándares (37°C y CO2 5%) crecen como taquizoitos; donde ocurren repetidos ciclos líticos en cualquier línea celular. Si estos parásitos son sometidos a estrés, se diferencian a bradizoitos. Si se anula el estrés ejercido, los bradizoitos vuelven a diferenciarse a taquizoitos imitando a lo que ocurre in vivo si falla el sistema inmune. Por lo tanto, la interconversión in vitro es una buena aproximación para el estudio de la diferenciación en T. gondii (Figura 1.3.2). Durante el pasaje de taquizoitos a bradizoitos, muchos genes se apagan y otros se encienden, permitiendo la fácil identificación de cada estadio (Figura 1.1.4); por ejemplo: SAG1 (antígeno de superficie) es específica de taquizoitos, mientras que BAG1 (antígeno de bradizoito) y LDH2 (lactato deshidrogenasa 2) son específicas de bradizoitos. Otra característica del pasaje a bradizoito es la formación de la pared quística, la cuál se puede evidenciar mediante el empleo de la lectina de Dolichos biflorus (DBL) la cual se une en la pared de los quistes (Figura 1.3.2). 15 )#$. Figura 1.1.4. Ciclo de vida en el hospedador intermediario (in vivo) y la diferenciación obtenida in vitro. En la fase aguda de la infección, el parásito se encuentra en estadio de taquizoito; mientras que en la fase crónica en bradizoito. El cámbio de estadio del parásito se debe a la presencia del sistema inmune in vivo y a diversos estrés in vitro. Los bradizoitos pueden volver a convertirse en taquizoitos si falla el sistema inmune o frente a la ausencia del estrés. Es posible identificar taquizoitos y bradizoitos debido a la expresión de proteínas específicas como SAG1 en taquizoitos y la pared del quiste (detectada con la lectina de Dolichos biflorus, DBL) en bradizoitos. Como se mencionó arriba, el pasaje de taquizoitos a bradizoitos in vitro ha sido asociado a cambios de temperatura, pH y otros inductores de estrés. Sin embargo, los mecanismos que utiliza el parásito para regular esta transición no se comprenden con claridad (para una revisión leer (Lyons et al. 2002). El conocimiento de estos mecanismos es de importancia central para establecer el control de la enfermedad. Varios estudios indican que el desarrollo iniciado tanto por esporozoitos como por bradizoitos para formar quistes es similar y pareciera ser la consecuencia de un programa genético unificado (Radke et al. 2003). Por otro lado, se obtuvieron mutantes químicas incapaces de diferenciar a bradizoitos, cuyo análisis genético reveló un orden jerárquico en la expresión de genes (Singh et al. 2002). En conjunto, estos resultados indican que el desarrollo del parásito se encuentra regulado a nivel transcripcional. 16 )#$. 1.5. Regulación de la transcripción Con el objetivo de definir la expresión de genes en T. gondii, Jerome y colaboradores desarrollaron el “Análisis Serial de la Expresión de Genes (SAGE)” (Jerome et al. 1998). Del análisis de estos experimentos se desprenden diversas conclusiones. Muchos transcriptos que codifican para proteínas del metabolismo basal y estructuras subcelulares parecen transcribirse sólo cuando se necesitan durante el crecimiento y desarrollo del parásito. Esto es consistente con el concepto de justo-atiempo descripto en Plamodium (Llinas and DeRisi 2004). Los genes específicos de desarrollo se encuentran distribuidos en todos los cromosomas. La organización de genes en clusters raramente ocurre y en esos casos, la expresión de los mismos no se encuentra influenciada por su proximidad física (Weiss and Kim 2007). El complejo basal de transcripción (clase II) que controla la expresión de genes codificantes en la mayoría de los organismos eucariotas es la ARN polimerasa II y los factores de transcripción generales (FTGs) asociados a ella. En Apicomplexas los FTGs se encuentran menos conservados que en eucariotas superiores. Sin embargo, estos parásitos presentan gran parte de la maquinaria basal de transcripción y de los factores remodeladores de cromatina requeridos para el control de este proceso (Meissner and Soldati 2005, Weiss and Kim 2007). Además, se observó que las regiones flanqueantes a las secuencias codificantes (CDS) actúan de forma autónoma. Esta propiedad, no solo le confiere una fuerza de expresión característica a cada CDS, sino que también, en ciertas situaciones le otorga una especificidad asociada al desarrollo; confirmando los principios básicos de una transcripción monocistrónica (Weiss and Kim 2007). Sin embargo, en ninguno de estos casos se logró definir claramente las secuencias que actúa en cis. Recientemente, Benhke y colaboradores (Behnke et al. 2008) identificaron dos elementos que actúan en cis en los promotores BAG1 y B-NTPasa, específicos de bradizoitos. En cada promotor se identificó un elemento diferente, al cual se unen proteínas diferentes. Dichos elementos fueron capaces de convertir un promotor constitutivo en uno cuya inducción se ve restringida a condiciones de cultivo de bradizoitos. A su vez, mediante un análisis in sílico, encontraron secuencias similares en regiones genómicas cercanas a genes importantes para la inducción a bradizoitos (LDH2, SAG4.2 y proteína de la pared del quiste de 65 kDa). Estos resultados confirman que la iniciación de la transcripción es el mecanismo principal que controla el desarrollo del parásito. Actualmente, se acepta que los factores de transcripción se unen y reclutan remodeladores de cromatina que descondensan la cromatina y facilitan el ensamblaje del complejo de preiniciación en las secuencias promotoras (revisado en (Li et al. 2004)). 17 )#$. Esto lleva a un nuevo enfoque de la regulación de la transcripción en la cual entra la dinámica de la cromatina y los mecanismos epigenéticos, en los cuales la pieza central son las histonas. 2. HISTONAS 2.1. Características generales Las histonas son el principal componente de la cromatina. Conforman una familia de proteínas básicas, de baja masa molecular, muy conservadas evolutivamente entre los eucariotas. Las cinco histonas mayoritarias, denominadas H1, H2A, H2B, H3 y H4, son proteínas ricas en aminoácidos (aa.) básicos cargados positivamente, los cuales son capaces de interaccionar con los grupos fosfatos del ADN, los cuales se encuentran cargados negativamente. Las cuatro histonas core o nucleosomales (H2A, H2B, H3 y H4) forman un octámero alrededor del cual se enrollan 146 pb. Este octámero se ensambla a partir de un tetrámero formado por dos histonas H3 y dos H4, al cual se agregan dos heterodímeros H2A-H2B (Figura 1.2.1). La histona externa o linker (H1) interacciona con el ADN internucleosomal. El conjunto del ADN enrollado alrededor del octámero de histonas, junto con la histona H1 y una cierta longitud de ADN internucleosomal constituye lo que se conoce como nucleosoma. Las histonas presentan un motivo estructural muy importante, denominado histone fold o pliegue de histonas. Consiste en una α-hélice corta, un giro o loop 1, una α-hélice larga, otro giro o loop 2 y otra α-hélice corta. Este motivo es el que permite que interaccionen entre si y formen el nucleosoma, desempeñando de este modo un papel decisivo en el primer nivel del empaquetamiento del ADN dentro del núcleo. Es importante resaltar que las regiones amino terminales (N-terminales) de las histonas quedan por fuera del nucleosoma y no presentan ninguna estructura determinada, por lo que reciben la denominación corriente de “cola” de la histona. Por muchos años se pensó que los nucleosomas eran partículas estáticas y que su función era meramente estructural. Ahora se sabe que los nucleosomas juegan un rol importante en la regulación de la transcripción y son capaces de transmitir información epigenética de una generación celular a la siguiente (Turner 2002). Se ha observado que si el nucleosoma se encuentra inmovilizado, previene la transcripción (Gottesfeld et al. 18 )#$. 2002). Además, que los nucleosomas presentan una actividad dinámica, la cual es dependiente de la secuencia del ADN y es facilitada por las modificaciones covalentes de las histonas. H2A H4 H2B H3 Dímero H2A-H2B Dímero H3-H4 Octámero de histonas Figura 1.2.1. Ensamblaje del octámero de histonas. Se forman dímeros H3-H4 y H2A-H2B. Dos dímeros H3H4 se unen para formar un tetrámero, el cual es base para la unión de dos dímeros H2A-H2B. Las regiones amino terminales de todas las histonas protruyen por fuera del octámero de histonas. © 2002 by Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walter. Tetrámero H3-H4 2.2. El lenguaje de las modificaciones covalentes de las histonas En el año 2000, Allis et al (Strahl and Allis 2000) propone la existencia de un lenguaje bioquímico capaz de modular la expresión de genes, al cual denomina código de histonas. Esta hipótesis fue creciendo para incluir la totalidad de los procesos que afectan la cromatina. Propone que (1) las diversas modificaciones postraducionales que ocurren en determinadas colas de las histonas pueden ser leídas por otras proteínas asociadas a cromatina, sirviendo como una plataforma para reclutar factores nucleares específicos, (2) distintas modificaciones en la cola de la misma u otra histona son interdependientes, originando varias combinaciones de distintas modificaciones en los mismos nucleosomas y (3) los distintos estados ordenados de la cromatina, como eucromatina y 19 )#$. heterocromatina, son dependientes de la concentración y combinación de nucleosomas diferencialmente modificados, generando un código epigenético (Escargueil et al. 2008). Las modificaciones postraduccionales de las histonas ocurren principalmente en las regiones N-terminales de las cuatro histonas nucleosomales, las cuales son flexibles y protruyen por fuera del nucleosoma. Estas incluyen acetilación, metilación, fosforilación, monoubiquitinación, citrulinación y ADP-ribosilación. Si bien es probable que la acetilación de las lisinas al neutralizar las cargas positivas y la fosforilación al agregar cargas negativas estén provocando la descondensación de la cromatina, el código de histonas es un mecanismo mucho más complejo. Se acepta que las histonas hiperacetiladas se encuentran mayormente asociadas a regiones genómicas activas; mientras que la desacetilación principalmente resulta en represión y silenciamiento. Por otro lado, la metilación presenta múltiples efectos en la función de la cromatina. Los residuos de lisina pueden ser mono-, di- o trimetilados, mientras los de arginina pueden ser mono- o dimetilados, ampliando el espectro de respuestas celulares posibles. Por ejemplo la metilación de H3 en lisina 9 (K9) está involucrada en silenciamiento y represión en muchas especies; mientras que la metilación de H3 en K4 se encuentra asociada con regiones de la cromatina activa. Además, la acetilación y la metilación pueden actuar de forma diferencial sobre la transcripción, dependiendo del residuo modificado. Por ejemplo, como ya vimos la metilación de H3 en K9 está asociada a silenciamiento, mientras que la acetilación del mismo residuo está asociado a transcripción activa (Fischle et al. 2003). Estas modificaciones pueden ser “leídas” por proteínas asociadas a cromatina, las cuales son las responsables de cumplir la función codificada por las histonas. Por ejemplo, las proteínas que presentan bromodominios son capaces de unir histonas acetiladas, promoviendo la activación de la transcripción. Estos bromodominios se encuentran en una amplia diversidad de proteínas como ser complejos remodeladores de cromatina y el factor de transcripción general TAF250. Otro ejemplo sería H3K9, que es capaz de ser reconocida por una proteína denominada HP1, encargada de mantener los dominios de heterocromatina. Por el contrario, ciertas modificaciones parecieran prevenir la unión de proteínas; como se observó en el caso de de metilación de H3 en K4, lo cual estaría inhibiendo el reclutamiento de HP1. Esto podría explicar en parte el sinergismo o antagonismo entre las distintas modificaciones. Probablemente las enzimas que modifican el mismo sitio se ven influenciadas por el estado de modificaciones que presenta la histona sustrato (Fischle et al. 2003). A diferencia de las histonas H3 y H4, las modificaciones postraduccionales de las histonas H2A y H2B prácticamente no se han estudiado. Probablemente debido a que los 20 )#$. dímeros de histonas H2A y H2B presentan una asociación más débil con el ADN nucleosomal y son frecuentemente desplazados de los nucleosomas; dando origen a la idea de que las modificaciones postraduccionales de estas histonas no serían tan importantes. Sin embargo, dos estudios realizados en levaduras, a las cuales se les deleciona la región N-terminal de H2A (Parra and Wyrick 2007) y de H2B (Parra et al. 2006), indican que éstas estarían reprimiendo la transcripción; ya que las mutantes presentan un gran aumento en la cantidad de genes encendidos. Esto es diferente a lo que ocurre con H3 y H4, cuyas regiones N-terminales afectan a la activación y represión de genes de forma balanceada. Esta comparación sugiere que los dominios de las histonas H2A y H2B poseen un rol mucho más importante en la regulación de la transcripción del que se les había otorgado previamente (Wyrick and Parra 2008). Otro nivel en el lenguaje de las histonas es la existencia de histonas variantes, las cuales son intercambiadas por histonas canónicas y se localizan de forma específica en los cromosomas. 2.2. Variantes y canónicas Las histonas variantes son formas no alélicas de las histonas convencionales. Se encuentran presentes en las células en muy baja cantidad comparada con los niveles de las histonas canónicas (Wu and Bonner 1981). La biosíntesis de las histonas canónicas, en eucariotas superiores, se encuentra restringida a la fase S, lo cual significa que ocurre simultáneamente con la síntesis de ADN y la replicación de los cromosomas. Mientras que las histonas variantes presentan una expresión independiente del ciclo celular. Mientras que H2A y H3 presentan numerosas variantes, H4 y H2B no presentan ninguna. Se ha observado que cada variante se encuentra involucrada en un proceso celular específico; indicando que el intercambio de histonas canónicas por variantes es uno de los mecanismos epigenéticos empleados para regular diferentes estados de la cromatina. La mayoría de los organismos eucariotas expresan una variante de H3 específica de centrómeros (cenH3), la cual se encuentra evolutivamente muy conservada en su parte globular, pero no en su región N-terminal (Malik and Henikoff 2003). Es esencial para la supervivencia de las células ya que presenta un rol fundamental en las funciones del centrómero durante la mitosis. La otra variante de H3 ampliamente distribuida en la naturaleza es la H3.3, la cual está asociada con la activación transcripcional. Las plantas, moscas, ranas y aves presentan además una H3.2 y sólo los mamíferos poseen aparte 21 )#$. una H3.1. Sin embargo estas últimas son expresadas e incorporadas en la cromatina de forma dependiente de la replicación (para mayor revisión ver (Hake and Allis 2006)). Por otro lado, la familia de las histonas H2A es la que presenta mayor número de variantes: H2AX, H2AZ, macroH2A y H2ABbd, las cuales se describen a continuación. 2.4. Variantes de H2A Las variantes de H2A difieren entre las especies: H2AZ y H2AX se encuentran prácticamente conservadas en todos los eucariotas, mientras que H2A-Bbd y macroH2A sólo se encuentran en vertebrados (Malik and Henikoff 2003, Kamakaka and Biggins 2005). H2AZ presenta menor divergencia evolutiva que el grupo de las H2A principal (Thatcher and Gorovsky 1994) y en parásitos presenta una región N-terminal más larga (Sullivan et al. 2006). Por otro lado, levaduras, Giardia lamblia, Entamoeba histolytica y Trichomonas vaginalis parecen haber reemplazado la H2A canónica por H2AX. Mientras que varios Apicomplexas, incluyendo Plasmodium ssp., no tienen H2AX, al igual que los Tripanosomátidos (Alsford and Horn 2004, Miao et al. 2006). H2ABbd presenta un 48% de identidad con la H2A canónica y es más corta. Recibe este nombre ya que se encuentra ausente en el cromosoma X inactivo (Barr body deficient). Además se encuentra asociada a la forma acetilada de H4 (Chadwick and Willard 2001) y los nucleosomas que contienen esta variante son menos estables que los que contienen la H2A canónica (Gautier et al. 2004); todo esto sugiere un posible rol en activación de la cromatina. Por el contrario MacroH2A se encuentra enriquecida en el cromosoma X inactivo en mamíferos de sexo femenino y en el corpúsculo XY transcripcionalmente silenciado en mamíferos de sexo masculino durante la meiosis. Estas también se encuentran en aves, las cuales presentan cromosomas sexuales diferentes que no experimentan inactivación del X. Recientemente, un análisis basado en microarreglos en células pluripotenciales humanas reveló que macroH2A se encuentra asociada a genes que codifican para reguladores que son claves para el desarrollo y el destino de esas células (Buschbeck et al. 2009). H2A.Z es esencial en la mayoría de los eucariotas superiores estudiados, salvo en levaduras. Su principal función ha sido otorgada a la activación de la transcripción, sin embargo se han descripto diversos mecanismos: (1) evitando la propagación de la heterocromatina en la eucromatina vecina (Meneghini et al. 2003) y (2) reclutando la ARN polimerasa II (Adam et al. 2001). Por otro lado, su región N-terminal es capaz de interaccionar con la proteína que une heterocromatina pericéntrica (INCENP), la cual es 22 )#$. crítica para la correcta segregación de los cromosomas (Rangasamy et al. 2003). Recientemente, se ha observado que también estaría cumpliendo una función en la reparación del ADN (Kalocsay et al. 2009). Como conclusión esta histona variante estaría involucrada en múltiples funciones, probablemente basadas en su capacidad para unirse a diversas proteínas reguladoras de diversos procesos nucleares. H2AX presenta gran homología con H2A canónica y se caracteriza por presentar el motivo carboxilo terminal (C-terminal) SQ(E/D)Ɏ, donde Ɏ es un aminoácido hidrofóbico. Esta variante juega un papel fundamental en la reparación del ADN frente a un quiebre de la doble hebra (DSB, del inglés double strands break); el cual se basa en la fosforilación de la S del motivo característico (Rogakou et al. 1998). El DSB es generado por agentes que dañan el ADN, pero también ocurre en varios mecanismos endógenos de la célula como meiosis y recombinación V(D)J. Si bien la fosforilación de esta histona se encuentra involucrada en todos estos procesos, es esencial sólo para la reparación del DSB. Brevemente, el daño del ADN recluta el complejo MRN compuesto por las proteínas Mre11-Rad50 y Nbs1 (Xrs2 en levaduras, por lo que el complejo se denomina MRX). Este es esencial para la activación de la kinasa ATM (ataxia telangiectasia mutada); la cual activa el arresto de la célula mediante la fosforilación de Chk2 (del inglés checkpoint kinase 2) y estimula la reparación mediante la fosforilación de H2AX. Sin embargo, las quinasas ATR (ATM relacionada a Rad3) y/o DNA-PK (ADN protein kinasa) también son capaces de fosforilar H2AX (Stiff et al. 2004). Existen dos mecanismos de reparación, la recombinación homóloga (RH) y la recombinación no homóloga (RNH). La H2AX fosforilada (γH2AX) participa en ambos tipos de reparación. Actúa como sitio de anclaje de varias proteínas, como los complejos INO80 y SWR1 a través de su unión a las subunidades Nhp10 y Arp4 de los mismos. En S. cerevisiae el complejo INO80 produce el desalojo de los nucleosomas que rodean el sitio de quiebre de la doble hebra, incluyendo nucleosomas que contienen γH2AX y H2AZ; ya que la presencia de nucleosomas en el sitio de reparación impide la asociación de las proteínas que facilitan el proceso. La figura 1.2.2 representa un esquema del modelo propuesto en levaduras. 23 )#$. Firura 1.2.2. Modelo de reparación del ADN en levaduras. El complejo Ku (formado por Ku70 y Ku80) y el complejo MRX (formado por Mre11, Rad50 y Xrs2) – MRN en humanos – se unen a los extremos del ADN. MRX recluta a las kinasas Mec1 (ATM) y Tel1 (ATR), las cuales fosforlilan a la H2AX cercana. La H2AX forforilada recluta a los complejos remodeladores SWR1 e INO80. SWR1 facilita la unión de Ku80, lo cual podría estimular la reparación por RNH (cuyas siglas en inglés son NHEJ); mientras que INO80 facilita la unión de MRX, lo cual estinularía la reparación por HR. INO80 remueve las histonas y facilita la reparación. Esto a su vez estimula el arresto de la célula hasta que la reparación sea resuelta (van Attikum H 2009). En ratones haploinsuficientes (H2AX (H2AX +/- ) o que carecen de una H2AX funcional -/- ), la eficiencia en la reparación de DSB se encuentra dramáticamente disminuida, el arresto G2/M inducido por daño fracasa y se observan numerosas anormalidades morfológicas, incluyendo defectos cromosómicos y mitóticos que provocan la infertilidad en machos (Bassing et al. 2002, Celeste et al. 2002, FernandezCapetillo et al. 2002). Recientemente, se ha mostrado que H2AX funciona, de forma dependiente de la dosis, como supresor de translocaciones oncogénicas y tumores en ratones (Bassing et al. 2003), mientras que en humanos es un marcador de regiones citogenéticas frecuentemente alteradas en diversos cánceres. Todos estos resultados, demuestran que H2AX presenta un rol vital en la identificación y reparación de genomas dañados. Por esto se la propone como la histona guardián del genoma (FernandezCapetillo et al. 2004). 24 )#$. 3. Lenguaje de las histonas en Toxoplasma 3.1. Evidencias de la presencia de mecanismos epigenéticos La epigenética (del griego epi, en o sobre) hace referencia, en un sentido amplio, al estudio de todos aquellos factores no genéticos que intervienen en el desarrollo de un organismo (ontogenia). Diversos estudios revelan que existen varios mecanismos diferentes para organizar y transmitir la información epigenética: interacciones entre el ADN y el nucleosoma, las cuales pueden ser moduladas por modificaciones postraducionales de las histonas dadas por enzimas específicas, intercambio de histonas canónicas por variantes, actividad de complejos remodeladores de cromatina los cuales requieren energía en forma de ATP, metilación o glucosilación de bases del ADN y localización subnuclear (Figura 1.3.1). Figura 1.3.1. Mecanismos epigenéticos (Epigenetic mechanisms). Sullivan et al (Sullivan 2006). Durante el desarrollo de esta tesis surgieron diferentes estudios que comenzaron a dilucidar la importancia de este mecanismo en el desarrollo de T. gondii. En el genoma del parásito, como en diversos parásitos protozoarios, se encontraron homólogos de casi todas las proteínas involucradas en estos mecanismos (Meissner and Soldati 2005, Sullivan and Hakimi 2006, Sullivan et al. 2006). 25 )#$. Al mismo tiempo se observó que en T. gondii, las regiones río arriba de genes expresados constitutivamente se encuentran enriquecidos en histonas H3 y H4 acetiladas tanto en taquizoitos y bradizoitos. En contraste, los genes específicos de bradizoitos se encuentran hipoacetilados en taquizoitos y se acetilan en condiciones de diferenciación in vitro. Del mismo modo, los genes específicos de taquizoitos se encuentran hiperacetilados en taquizoitos y este nivel de acetilación disminuye bajo condiciones de diferenciación a bradizoitos. Otra modificación postraduccional estudiada es la metilación de H3 en K4. Esta puede estar di- y/o trimetilada; la dimetilación se encontró en genes activos e inactivos, mientras que la trimetilación está presente exclusivamente en genes activos (Saksouk et al. 2005). La acetilación y metilación de las histonas del parásito se correlaciona con la expresión de genes específicos de estadio. En taquizoitos, un complejo de histonas desacetilasas (HDAC) denominado TgCRC opera en promotores específicos de bradizoitos, mientras que la histona acetil transferasa (HAT) denominada TgGCN5, lo hace en promotores específicos de taquizoitos (Saksouk et al. 2005). TgGCN5 presenta una preferencia por la lisina 18 de la histona H3. En humanos esta modificación se encuentra mediada por la HAT denominada p300, la cual recluta una arginina metilasa, CARM1, para activar la transcripción. En Toxoplasma se caracterizó el complejo TgCARM1, el cual metila la arginina 17 y actúa en concierto con la acetilasa de histonas TgGCN5; por lo que la histona H3 se encuentra metilada en R17 y acetilada en la K18 en el mismo loci. Consecuentemente, la transcripción estaría siendo regulada mediante la asociación de HDAC y HAT y la subsecuente acetilación y desacetilación de histonas en genes específicos de estadio. A su vez, la inhibición de CARM1 por un inhibidor específico induce la diferenciación de taquizoitos a bradizoitos, indicando que el ciclo de vida del parásito puede ser manipulado interfiriendo en la maquinaria epigenética (Saksouk et al. 2005). Figura 1.3.2. Modelo del control de la expresión génica en la interconversión. Tachyzoite (taquizoito), Bradyzoite (bradizoito), ON (encendido), OFF (apagado), Ac (acetilación), Me (metilación). Fuente: Saksouk N et al 2005. 26 )#$. Apoyando estos resultados, Gissot y colaboradores encontraron que las regiones 5’ de todos los genes predichos del cromosoma Ib, están enriquecidas con las siguientes modificaciones postraduccionales: AcH4 (H4 acetilada), H3K9ac (H3 acetilada en K9) y H3K4me3 (H3 trimetilada en K4). Dichos enriquecimientos comprenden aproximadamente 700 nucleótidos. La distancia entre el pico de AcH4 y H3K9ac y el codón de inicio es menor a 1.000 pb en más del 85% de los genes y es menor a 1.500 pb para más del 90%. De modo similar, el pico de H3 dimetilada en R17 termina aproximadamente a 1.000 pb del codón de iniciación; aunque el enriquecimiento de esta modificación se encontró sólo en el 4,5% de los genes analizados. Es importante resaltar que el pico de H3K4me3 se encuentra desplazado hacia el codón de inicio en comparación con AcH4; permitiendo predecir la dirección de transcripción de los promotores en T. gondii (Gissot et al. 2007). Figura 1.3.3. Distribución genómica de modificaciones postraduccionales. Esquema que representa una región del cromosoma Ib. En celeste se encuentran indicados los CDS; en azul los picos de AcH4 y en rojo los enriquecimientos observados para H3K4me3. La línea punteada indica el final del pico de H3K4me3 con respecto al CDS. Fuente: Gissot et al (2007). Por otro lado, en Toxoplasma y P. falciparum se describió la presencia de la histona metil transferasa (HMT), Set8, la cual tiene la capacidad de mono-, di-, y trimetilar la K20 de la histona H4; a diferencia de su ortólogo en humanos que sólo es capaz de monometilar K20. Esta enzima se encuentra regulada a nivel del ciclo celular y se encuentra enriquecida en la heterocromatina pericéntrica y telomérica en ambos parásitos, al igual que sus productos (la histona H4 mono- y dimetilada en K20) y la histona H3 trimetilada en K9 (marcador de heterocromatina en todos los eucariotas superiores estudiados) (Sautel et al. 2007). Con respecto a la presencia de histonas variantes, solo se describió la presencia de una H3 variante, la H3.3. Esta presenta solo cuatro aminoácidos diferentes con respecto a la H3 canónica (Sullivan 2003). Como se puede observar, la mayoría de los mecanismos epigenéticos se encuentran conservados en T. gondii; sin embargo, todos los estudiados hasta el momento tienen a las histonas H3 y H4 como protagonistas. Las histonas H2A y H2B 27 )#$. también están involucradas en diversos procesos celulares y las variantes de H2A varían en las distintas especies. Por lo que nuestra hipótesis es que Toxoplasma presenta histonas H2A y H2B características, involucradas en diversos mecanismos epigenéticos. 28 )#$. OBJETIVOS: General: El objetivo general de esta tesis es conocer en mayor detalle los mecanismos epigenéticos que involucran a las histona H2A y H2B en T. gondii. Esto permitirá conocer las vías que utiliza el parásito para regular su crecimiento y diferenciación a otros estadios. Específicos: 1. Identificar las histonas H2B de T .gondii, describir similitudes y diferencias entre ellas, identificar la presencia de variantes y analizar su expresión en los estadios de taquizoito y bradizoito. 2. Identificar las histonas H2A de T .gondii, describir similitudes y diferencias entre ellas, identificar la presencia de variantes y analizar su expresión en los estadios de taquizoito y bradizoito. 3. Estudiar la composición de los nucleosomas a través de la interacción de las histonas H2A y H2B. El estado de acetilación y/o su proximidad a histonas acetiladas; como también su localización en el genoma. 29 Capítulo 2 )#$ 2. 1. Introducción En mamíferos las histonas H2B pertenecen a una familia multigénica (Albig et al. 1999). Presenta dos linajes: uno que incluye un inmenso número de copias de H2Bs canónicas y otro que contiene dos histonas particulares: hTSH2B (Zalensky et al. 2002) y H2BFWT (Boulard et al. 2006). Estas últimas, son tejido específicas, las cuales se encuentran en gametas y no se conocen sus funciones. Por lo tanto, se asume que esta familia, al igual que la familia de las histonas H4 no posee variantes (Malik and Henikoff 2003). Las H2Bs poseen una hélice Į en la región C-terminal además del plegamiento característico de histonas. Esta Į-hélice de cada H2B presente en un nucleosoma se ubica en los planos exteriores del mismo y podría interaccionar con otras unidades nucleosomales generando una estructura de orden superior. A su vez, las histonas H2Bs poseen una cola N-terminal, la cual se determinó que es crucial para la ensamblado de los cromosomas mitóticos (de la Barre et al. 2000, de la Barre et al. 2001). Dentro de las células, estas histonas se encuentran formando dímeros con histonas de la familia H2A como componentes de los nucleosomas. Sin embargo, existe un grupo de dímeros solubles, los cuales pueden ser intercambiados con los que están en la cromatina por complejos modeladores de cromatina. El intercambio de dímeros H2AZ-H2B por el complejo SWI/SNF es el caso más estudiado (Mizuguchi et al. 2004). Estudios in vitro demostraron que este complejo también es capaz de desalojar y/o incorporar tanto la H2B canónica como las variantes hTSH2B y H2BFWT (Boulard et al. 2006). Si bien el código de histonas de las H2Bs se encuentra poco caracterizado, se han descripto modificaciones postraduccionales características y particulares. En levaduras, la histona H2B es capaz de mono-ubiquitinarse en la K123 (K120 en humanos), promoviendo dos funciones diferentes. Por un lado reclutaría metiltransferasas (como Dot1 y Set1 en levaduras) promoviendo la trimetilación de histonas H3 (en K4 y K79 en levaduras) durante la elongación en el proceso de transcripción (Xiao et al. 2005). Por otro lado, estaría reclutando el complejo SAGA mediante su unión a Ubp8 (proteasa específica de ubiquitina que forma parte del complejo SAGA). Este última deubiquitina las H2B, lo cual es requerido para el inicio de la transcripción; al menos de los genes regulados por este complejo (Henry et al. 2003, Daniel et al. 2004). Parra y colaboradores encontraron que la deleción de la región N-terminal de H2B en S. cerevisiae estimula la expresión de una inmensa cantidad de genes, indicando un 31 )#$ mayor aporte de esta región al proceso de silenciamiento (Parra et al. 2006). Recientemente se había descripto que H2B era capaz de sumoilarse en los residuos K6/K7 y K16/K17, lo cual es requerido para el silenciamiento en los telómeros y algunos genes localizados en eucromatina (Nathan et al. 2006). Sin embargo, solo la deleción de los residuos 30 a 37 es la responsable del fenotipo observado previamente; por lo que se lo denominó dominio represor de la histona H2B: “dominio HBR” (del inglés “histone H2B repressive”). Éste es requerido para la represión del 8,6% del genoma de levaduras, incluyendo genes involucrados en el metabolismo de vitaminas y carbohidratos localizados en regiones teloméricas y subteloméricas (Parra et al. 2006). Otra modificación postraduccional característica, es la fosforilación de la H2B S10 en levaduras (S14 en mamíferos). Esta ocurre durante la compactación de la cromatina en el proceso de apoptosis (Ahn et al. 2005). Resulta muy interesante el hecho de que esta fosforilación, mediada por la kinasa Ste20, se encuentra regulada por el estado de acetilación de la lisina vecina. La inducción de la apoptosis promueve la desacetilación de H2B K11 por Hos3, lo cual permite la fosforilación de H2B S10 y la condensación de la cromatina (Ahn et al. 2006). La mutación de todas las lisinas de la región N-terminal demostró que las acetilaciones de H2B en levaduras tienen un rol en la activación de la transcripción (Parra et al. 2006). Al momento de inicio de este trabajo, no se conocía mucho a cerca de las histonas de T. gondii. Se habían identificado dos histonas H3 (H3 y H3.3) las cuales muestran algunas características peculiares a nivel de secuencia (Sullivan 2003) y se había clonado un gen de la familia h2b, el cual fue utilizado para estudiar la división nuclear de los parásitos in vivo al ser fusionado a la proteína fluorescente amarilla (Hu et al. 2004). En T. brucei y P. falciparum se habían determinado que los niveles de ARNm de las H2Bs se encontraban regulados de forma diferente a lo largo del ciclo celular como también de los distintos estadios de diferenciación (Garcia-Salcedo et al. 1999, Lobo and Kumar 1999). Basados en estos antecedentes decidimos identificar las histonas H2B de T .gondii, describir similitudes y diferencias entre ellas, identificar la presencia de variantes y analizar su expresión en los estadios de taquizoito y bradizoito. 32 )#$ 2. 2. Resultados 2. 2. 1. Identificación y clonado Haciendo uso de la información y programas presentes en las bases de datos del National Center for Biotechnology Iinformation (NCBI: http://www.ncbi.nlm.nih.gov/) y de la base de datos de T. gondii (ToxoDB: http://toxodb.org), se identificaron 3 histonas H2B putativas en este parásito. Estas fueron denominadas H2Bv, H2Ba y H2Bb en base al estudio filogenético explicado a continuación (ver sección 2.2.2). Se clonaron mediante la técnica de PCR utilizando ADNc y ADNg como templado. La secuencia codificante (CDS) de H2Bv codifica para una proteína de 124 aa con una masa molecular predicha de 13,7 kDa y punto isoeléctrico pI= 10,5 (AN: AF406555). La CDS de H2Ba codifica para una proteína de 117 aa con masa molecular de 12,6 kDa y pI=10,1(AN: DQ118972). Mientras que la CDS de H2Bb codifica para una proteína de 115 aa con una masa molecular de 12,5 kDa y pI=10. Mediante la utilización del programa ToxoDB Genome Browser (http://toxodb.org) se pudo observar que las tres cepas de parásitos secuenciadas, tipo I (GT1), tipo II (Me49) y tipo III (VEG), poseen las tres histonas H2B. A su vez, cada una se encuentra en sintenia: H2Bv se localizada en el cromosoma Ib (TGGT1_022960, TGME49_009910, TGVEG_038620), H2Ba en el cromosoma IX (TGGT1_042340, TGME49_105160, TGVEG_106580) y H2Bb en el cromosoma XII (TGGT1_103250, TGME49_051870, TGVEG_000470). También se analizó la presencia de Polimorfismo de un solo nucleótido (SNPs) para cada una de estas histonas en cepas de los tres tipos. Se pudo observar que los cambios en la secuencia de H2Bv ocurren solo en regiones no codificantes, dando de este modo una densidad de SNPs a lo largo de la secuencia codificante nula (Tabla 2.2.1). En los casos de las histonas H2B canónicas se observa polimorfismo de una base en la secuencia codificante, aunque en muy baja densidad: H2Ba contiene 1 SNP y H2Ba contiene 2. Además, estos cambios son sinónimos, no alterando de este modo la composición ni la función de la proteína (Tabla 2.2.1). La ausencia de SNPs estaría indicando una selección purificadora del gen, lo cual está relacionado con la importancia de conservar estas proteínas. 33 )#$ Tabla 2.2.1. Análisis de SNPs. Cepas Densidad SNPs SNPs Gen comparadas en CDS Totales No sinónimos Sinónimos No codificante H2Bv ME49 vs RH ME49 vs VEG GT1 vs ME49 0 0 0 5 5 3 0 0 0 0 0 0 5 5 3 H2Ba GT1 vs ME49 GT1 vs VEG 2,85 2,85 1 1 0 0 1 1 0 0 H2Bb GT1 vs ME49 GT1 vs VEG 5,75 5,75 2 2 0 0 2 2 0 0 La composición aminoacídica de las 3 H2Bs fue analizada mediante un alineamiento de la secuencia de proteína y un análisis de la estructura secundaria de las mismas (Figura 2.2.1). La secuencia de aa de H2Ba y H2Bb presentan un 94% de homología entre ellas; mientras que H2Bv presenta un 68% de identidad (82% similitud) con H2Ba y H2Bb. La mayor homología de secuencia se observa en la región que comprende de la α-hélice 1 a la 3, la cual corresponde al histone fold o pliegue de histonas. En la región carboxilo terminal (C-terminal) se encuentra una α-helix (αC) característica de las histonas H2B, la cual contiene un lisina (Lys; K) conservada susceptible a ser monoubiquitinada (Luger et al. 1997). 1 MSGKGPAQKSQAAKKTAGKSLGPRYRRRKRTESFALYIYKVLKQVHPETGVSKKSMSIMN 1 MVAKKSA-KS-AKPKASGKS-GK--GKKKRAESYSSYIFKVLKQVHPETGISKKSMMIMT 1 MVAKKSA-KV-AKSKDSGKK-GK--GKK-RAESYSSYIFKVLKQVHPETGISKKSMMIMT * .* .* ** * *::*** * ::**:**:: **:***********:***** **. H2Bv H2Ba H2Bb L1 α1 Ub 71 SFINDIFDRLADEAVRLIRYNKKRTLSSREIQTAVRLLLPGELSKHAVSEGTKAVTKYTTSGA 66 SFIADTFDKIASEAGKLCKYNKKDTLSSREIQTAVRLVLPGELAKHAVSEGTKAVTKYTGK 65 SFIADTFDKIATEAGKLCKYNKKDTLSSREIQTAVRLVLPGELAKHAVSEGTKAVTKYTGK *** * **::*.** :* :**** *************:*****:*************** . H2Bv H2Ba H2Bb L2 α2 α3 αC Figura 2.2.1 Alineamiento de las secuencias de aminoácidos de las H2B de Toxoplasma. Los aminoácidos idénticos están marcados con (*) y sombra gris; los aminoácidos homólogos se encuentran marcados con (:) o (.) dependiendo de cuán cercanos sean y aminoácidos heterólogos se encuentran marcados con un espacio en blanco. Debajo, se encuentra una representación esquemática de la estructura secundaria donde α y L significan α-hélice y loop, respectivamente. 34 )#$ Por otro lado, la mayor divergencia se encuentran principalmente en la región Nterminal. Esta región se caracteriza por ser susceptible a modificaciones postraduccionales como acetilación (Ac), metilación (Me) y fosforilación (P). Tal como puede verse en la figura 2.2.2, estas histonas poseen varios residuos K, arginina (R) y serina (S), los cuales podrían ser modificados postraduccionalmente. H2Ba y H2Bb poseen mayor número de residuos K que H2Bv. Por el contrario, H2Bv posee varios residuos de argininas (R24, R26, R27 and R28) que están ausentes en H2Ba y H2Bb. Todas presentan varios residuos S: H2Bv presenta cuatro (S1, S10, S20 y S33) mientras que H2Ba presenta siete (S6, S9, S15, S18, S28, S30 and S31). Se han descripto varias modificaciones postraduccionales en la región N-terminal de S. cerevisiae (ScH2B) y humanas (HuH2B). Sin embargo, resulta difícil deducir los residuos de las histonas H2B de Toxoplasma que estarían modificados, debido a la divergencia existente entre éstas especialmente en la región N-terminal (Figura 2.2.2). Plasmodium spp. también posee una H2B canónica y una H2Bv (Figura 2.2.3 y (Miao et al. 2006)) Recientemente se describieron modificaciones postraduccionales para las histonas H2B de P. falciparum (PfH2B y PfH2Bv) (Trelle et al. 2009). En este caso observaron que la histona PfH2Bv posee múltiples acetilaciones en la región N-terminal, mientras que en la canónica solo encontraron la ubiquitinación en la región C-terminal. Todos los residuos acetilables de PfH2Bv se encuentran en TgH2Bv (Figura 2.2.2), por lo que se podría pensar que en Toxoplasma podría ocurrir lo mismo. En S. cerevisiae se describió un dominio HBR, el cual es requerido para la represión del 8,6% de su genoma. Este se encuentra subrayado en la Figura 2.2.2. Se determinó que los aa. requeridos para esta función son la lisina en posición 34 (K34) y una lisina flanqueante (K30 o K37) (Parra et al. 2006). Si bien, por homología de secuencia, se observó que este dominio se encuentra conservado en varias especies (Parra et al. 2006), no se encuentra conservado en ninguna de las H2B de T. gondii. Sin embargo, H2Ba y H2Bb contienen en esa región varios residuos de lisina, los cuales tendrían otra lisina ubicada a 3 o 5 aa. río arriba; mientras que H2Bv no tiene ninguna de ellas (Figura 2.2.2). 35 )#$ M Fosforilación TgH2Ba TgH2Bb PfH2B HuH2B ScH2B M Acetilación α1 MVA---KKSAK---------SAKPKA-----SGKSGKGKKKRAESYSS MVA---KKSAK---------VAKSKD-----SGKKGKGKK-RAESYSS MVS---KKPAK---------AKKTGTGP---DGKK-KRKKSRYDSYGL MPEPAKSAPAPKK------GSKKAVTKAQKKDGKK--RKRSRKESYSV MSSAAEKKPASKAPAEKKPAAKKTSTSV---DGKK--RSKARKETYSS YIFKVLKQVHPETGI YIFKVLKQVHPETGI YIFKVLKQVHPDTGI YVYKVLKQVHPDTGI YIYKVLKQTHPDTGI dominio HBR α1 TgH2Bv PfH2Bv ScH2B MS--------GKGPAQKSQAAKKTAGKSLGPRYRRRKRTESFAL MS--------GKGPAQKSQAAKKTAGKSLGPRXRRKXRTESFSL MSSAAEKKPASKAPAEKKPAAKKTS-TSVDGKKRSKARKETYSS YIYKVLKQVHPETGV YIYKVLKQVHPETGV YIYKVLKQTHPDTGI dominio HBR Figura 2.2.2. Alineamiento de la región N-terminal de las H2B. En el esquema superior comparan las H2B canónicas de Toxoplasma (H2Ba y H2Bb) y P. falsiparum (PfH2B), la secuencia consenso de las H2B humanas (HuH2B) y la H2B de S. cerevisiae (ScH2B); mientras que en el esquema inferior se comparan las H2B variantes de Toxoplasma (H2Bv) y P. falsiparum (PfH2B) y la H2B de S. cerevisiae (ScH2B). Los aminoácidos modificados posttraduccionalmente se encuentran sombreados; el color de la sombra denota la modificación de acuerdo a lo establecido en la parte superior del esquema. HBR domain: dominio represor de histona H2B. Las flechas indican las lisinas importantes del dominio HBR. El cilindro indica los aa correspondientes a la hélice α1. 2. 2. 2. Análisis filogenético Dado que T. gondii presenta varias H2B, se buscaron secuencias de H2Bs de los parásitos del phylum Apicomplexa presentes en las bases de datos y se las comparó por análisis de Neighbor joining (NJ), incluyendo las secuencias de humanos. Los resultados muestran claramente que existen dos clados diferentes de H2B en cada una de las especies analizadas (Figura 2.2.3). En uno de los grupos se encuentra TgH2Bv y en el otro TgH2Ba y TgH2Bb. En paralelo a nuestro trabajo, se caracterizaron las histonas H2B de P. falciparum (Miao et al. 2006) y se las nombró como PfH2B (mayor homología con HuH2B) y PfH2B variante (PfH2Bv). Basados en esta nomenclatura se denominaron como H2B variantes a todas las histonas que se agrupan con PfH2Bv. Las secuencias de las H2B del grupo de las variantes muestra mayor homología entre ellas que las del grupo de las canónicas. Las diferencias se encuentran principalmente en la región Nterminal. 36 )#$ Figura 2.2.3. Árbol de NJ construido con de secuencias de H2B de varios miembros del filum Apicomplexa. Tg: T. gondii (H2Bv1: AF406555, H2Ba: DQ118972, H2Bb: TGME49_051870), Pf: Plasmodium falciparum (H2Bv: BU497652, H2B: U14734), Pv: Plasmodium vivax (H2B: CAA76839) Et: Eimeria tenella (H2Bv1: CD345163; H2B: CD569058), Nc: Neospora caninun (H2Bv1: CF598462; H2B: CF937965), Sn: Sarcocystis neuron (H2Bv1: H2B: CV194457), Ch: CV193312; Cryptosporidium hominis (H2Bv1: XP_666723, H2B: XP_666188). hTSH2B: human testis/sperm specific H2B: AP397301. Hu: human (H2B: secuencia consenso). 2. 2. 3. Antigenicidad de H2Bv en pacientes con toxoplasmosis Las histonas H2Bs fueron caracterizadas en Plasmodium debido a su valor antigénico, ya que los pacientes infectados presentan serología positiva para esta histona (Rawat et al. 2004). Por lo tanto, se analizó el valor antigénico de la H2Bv de T. gondii. Para ello, se evaluó la reactividad de sueros humanos de individuos seronegativos y seropositivos con infección crónica y aguda por ELISA utilizando placas sensibilizadas con H2Bv recombinante (rH2Bv). Esta proteína se obtuvo mediante la expresión en bacterias y purificación con columna de niquel (Figura 2.2.4A). Ninguno de los sueros fue reactivo para esta histona (datos no mostrados). 2. 2. 4. Obtención de anticuerpos específicos Tanto H2Bv como H2Ba fueron expresadas como proteínas recombinantes (rH2Bv y rH2Ba, respectivamente). Ambas fueron clonadas en vectores de expresión en bacterias, expresadas mediante la inducción con IPTG y purificadas en columnas de niquel como se describe en el capítulo 6. Ambas proteínas recombinantes se expresaron de forma soluble, las cuales migraron con una masa molecular aparente de aproximadamente 17 kDa en SDS-PAGE 15% (Figura 2.2.4A). Con el fin de obtener anticuerpos específicos para cada una de estas histonas, se inmunizaron dos ratones con rH2Ba y un conejo con rH2Bv. Sin embargo, solo se obtuvo suero policlonal reactivo para rH2Bv (α-H2Bv). La especificidad de este anticuerpo fue determinada por Western blot, 37 )#$ donde se observa que es muy específico para rH2Bv y presenta una débil reacción cruzada con rH2Ba (Figura 2.2.4B). Al analizar la afinidad de este anticuerpo por las dos H2Bs recombinantes mediante ELISA competitivo, se observó que α-H2Bv pierde la capacidad de unirse a rH2Bv inmovilizada, cuando es preincubado con la misma proteína, incluso a concentraciones muy bajas como 0,1 μg/ml. Mientras que preincubar el anticuerpo con rH2Ba o rROP2 (proteína recombinante de T. gondii no relacionada con histonas) no afectan significativamente la interacción entre α-H2Bv y la rH2Bv inmovilizada, incluso a concentraciones 5 μg/ml (Figura 2.2.4C y datos no mostrados). Figura 2.2.4. Anticuerpo antiH2Bv. A. Las proteínas recombinantes H2Ba y H2Bv purificadas y analizadas por SDS-PAGE 15%, utilizadas para la obtención de anticuerpos. B. La especificidad y reacción cruzada de α-H2Bv examinada por Western blot, utilizando rH2Ba y rH2Bv. La membrana teñida con Ponceau, permite visualizar la cantidad presente de cada pretína recombinante. C. ELISA competitivo que muestra la afinidad de α-H2Bv por distintas proteínas recombinantes. Se evaluó la capacidad de α-H2Bv (1/10.000) preincubado con 0; 0,1; 0,5 y 1 μg/ml de rH2Bv (), rH2Ba () y rROP2 (•) de reconocer la proteína inmobilizada (rH2Bv). 2. 2. 5. Inmunodetección de las H2Bs Con el objetivo de caracterizar las histonas H2B de Toxoplasma a nivel de proteína, se realizó un Western blot con α-H2Bv de extracto total de taquizoitos y bradizoitos obtenidos in vitro y células hospedadoras (HFF) como control. Se detectaron dos bandas de masas moleculares aparentes de aproximadamente 15 y 17 kDa (Figura 2.2.5). Las cuales no se observan en el extracto total de células control, indicando que 38 )#$ son bandas específicas de H2B del parásito. El suero preinmune fue no reactivo en todos los casos (datos no mostrados). Estas dos bandas podrían ser dos estados postraduccionales diferentes de H2Bv. Sin embargo, no se puede descartar la posibilidad de que exista algo de reacción cruzada con H2Ba y las dos bandas corresponden a las dos histonas H2B. Además, se comparó la intensidad de las bandas detectadas con α-H2Bv en ambos estadios (Figura 2.2.5). La cantidad de proteína correspondiente a cada estadio fue evaluada mediante Western Blot con anti-actina de cantidades decrecientes (20, 15, 10 and 5 μl) de cada muestra. En la Figura 2.2.5 se muestra el Western Blot correspondiente a 5 μl de cada muestra, donde la reacción de color no se encuentra saturada. Se podría concluir que los niveles de H2B son iguales en ambos estadios. Figura 2.2.5. Identificación de H2Bs en taquizoitos (Tz) y bradizoitos (Bz) por Western blot (WB). Se extrajeron las proteínas correspondientes a taquizoitos (Tz), bradizoitos (Bz) in vitro y células hospedadoras (HFF) utilizando TRIZOL (Invitrogen). El porcentaje de diferenciación a Bz fue de 87%, analizado mediante la detección de la pared del quiste con la lectina de Dolicos biflorus fluorescente. 20 μl de cada muestra fue separada por electroforesis en SDS-PAGE 18% y analizada por WB con α-H2Bv; mientras que 5 μl fueron analizados con anti-actina (α-Actin) para cuantificar la cantidad de proteína utilizada. Por otro lado, se determinó la localización de esta proteína dentro del parásito por inmunofluorescencia indirecta (IFI). Esta se llevó a cabo en parásitos intracelulares, crecidos en condiciones de taquizoito y bradizoito in vitro, fijados con paraformaldheído y permeabilizados con 0,2% Tritón X-100. Se detectó señal sólo en el núcleo, excluyendo el nucléolo, en ambos estadios (Figura 2.2.6). 39 )#$ Figura 2.2.6. Inmunofluorescen ncia Indirecta. Se realizó en taquizoitos (Tz) y bra adizoitos (Bz) in vitro. Con el fin de identificar los distintos d estadios se utilizó anti-SAG1 (α-SAG1) com mo marcador de taquizoitos y anti-p21 (α-p21) com mo marcador de bradizoitos. La presencia de H2Bv en e el núcleos se analizó superponiendo su marca con DAPI. Se seleccionaron dos zonas para ser o observadas con mayor aumento, las cuales se mu uestran en el centro de la figura. Una de las zonas ccorresponde a la señal observada con α-H2Bv en Tz T y la otra en Bz (cuadrados blancos). Las flechas blancas indican la ausencia de señal en los nucleo olos. 2. 2. 6. Análisis de his stonas purificadas Las histonas de Toxo oplasma se purificaron mediante una extracción con ácido sulfúrico y luego precipitadas por toda noche con acetona en frío. Estas fuerron separadas por SDS-PAGE 15% y analiz zadas por Western blot con α-H2Bv. Dos ban ndas de masa molecular aparente menor a H2AX (variante de la histona H2A, utilizada como c control) 2.7A). fueron detectadas (Figura 2.2 Para obtener una mejor m resolución, las histonas purificadas fueron luego separadas por relación ca arga/masa en electroforesis en geles de poliacrilamida conteniendo tritón-ácido acéttico y urea (TAU-PAGE). Para identificar cada histona, se cortaron varias bandas, las cu uales fueron enviadas para su análisis por espe ectrometría de masa. Se identificaron las cu uatro histonas encargadas de formar el nucleo osoma (Figura 2.2.7B). Varias bandas fue eron identificadas como H4, basados en el patrón que 40 )#$ presentan, podría pensarse que se deben a versiones modificadas postraduccionalmente de esta histona. Sólo una banda fue identificada como H3. Si bien se detectaron tres bandas correspondientes a H2A, no fue posible identificar cada una debido a que los péptidos obtenidos eran iguales en todos los casos. Finalmente, se identificaron dos bandas H2B, ambas correspondientes a H2Bv (Figura 2.2.7B). Un ensayo de Western blot con α-H2Bv de histonas extraídas y separadas en las mismas condiciones, reveló dos bandas; las cuales coinciden con las dos bandas identificadas por MALDI-TOF. En base a estos resultados nos inclinamos a pensar que las dos bandas corresponden a H2Bv y que ésta sería la histona H2B mayoritaria, dado que H2Ba no fue identificada en ninguna de las bandas analizadas por espectrometría de masa. Figura 2.2.7. Análisis de histonas purificadas. A. Histonas separadas por SDS-PAGE 15% y transferidas. La membrana fue teñida con Ponceau para visualizar las bandas y dividida a la mitad. Ambas fueron analizadas por WB, una con α-H2Bv y la otra con α-H2AX como control. B. Histonas purificadas analizadas por TAU-PAGE. Una muestra fue teñida con Coomassie blue y las bandas marcadas con números fueron cortadas y analizadas por espectrometría de masa. La identidad de las bandas se encuentra indicada al lado de cada número. Otra muestra fue transferida y analizada por Western blot (WB) utilizando α-H2Bv. 2. 2. 7. Expresión de las histonas H2Bs de T. gondii Con el objetivo de estudiar la expresión de las tres histonas H2Bs, se realizaron ensayos de PCR con oligonucleótidos específicos para cada una de ellas utilizando como templado ADNg y ADNc de taquizoitos y bradizoitos. Todas fueron correctamente amplificadas de ADNg, corroborando de este modo la presencia de ellas en el genoma del parásito (Figura 2.2.8). Cuando se determinaron los niveles de transcripto de estas histonas, se observó que h2bv y h2ba se expresan tanto en taquizoitos como en bradizoitos, mientras que h2bb no se transcribe en estos estadios o lo hace en muy baja cantidad, ya que no pudo ser amplificada por esta metodología (Figura 2.2.8). Este último resultado se encuentra avalado por la ausencia de ESTs correspondientes a h2bb en la base de datos ToxoDB. 41 )#$ ARNm Tz Bz ADNg Figura 2.2.8. Amplificación de las CDS de h2bb, h2ba y h2bv por PCR utilizando como templado ADNg y ADNc obtenido de taquizoitos (Tz) y bradizoitos (Bz). La visualización de las mismas se llevó a cabo por electroforesis en gel de agarosa 1% y tinción con bromuro de etidio. h2bb h2ba h2bv Dado que h2bv y h2ba son las dos histonas H2B presentes en los estadios analizados y basados en que las histonas variantes por lo general son las histonas minoritarias, nos propusimos comparar la cantidad de transcripto correspondiente a cada una de ellas en el estadio taquizoito. Para ello, se compararon las eficiencias de las reacciones de PCR utilizadas para amplificar cada histona utilizando ADNg como templado, ya que estos genes son de copia única. Se observó que ambas curvas coincidían, indicando que las eficiencias de amplificación de ambas PCRs son muy similares (Figura 2.2.9B). A continuación se llevaron a cabo ensayos de RT-PCR semicuantitativa, utilizando ADNc de taquizoitos como templado. De este modo, se comprobó que los niveles de expresión de h2bv son significativamente mayores que los de h2ba (Figura 2.2.9A). Figura 2.2.9. Niveles de transcripto de H2B en taquizoito. A. PCR semicuantitativa utilizando ADNc como templado. B. PCR semicuantitativa utilizando ADNg como templado. En la parte superior de la figura se obserban los productos de amplificación para cada número de ciclo en un gel de agarosa 1%. En la parte inferior, se encuentra el gráfico de la densidad óptica integrada (IOD) de cada una de las bandas con respecto al número de ciclos. 42 )#$ A su vez, se comparó la expresión de estas dos histonas en los estadios taquizoito y bradizoito in vivo. Los taquizoitos son altamente replicativos, mientras que los bradizoitos de quistes de cerebro de ratón se encuentran mayoritariamente arrestados en la fase G0. Como las histonas canónicas se transcriben en la fase S del ciclo celular (Harris et al. 1991) y las histonas variantes lo hacen de forma independiente, al comparar estos estadios estaríamos comparando, de forma indirecta, la expresión de las histonas con el ciclo celular. Los niveles de transcripto de h2bv y h2ba se determinaron por RTPCR semicuantitativa con ADNc de taquizoitos y bradizoitos in vivo. Como control de carga se utilizó la amplificación del gen de α-tubulina. En estas condiciones, los niveles de transcripto de h2ba se encuentran disminuidos en bradizoitos con respecto a taquizoitos; mientras que los de h2bv se mantienen constantes en ambos estadios (Figure 2.2.10). Tz Tz Q Q h2bv h2ba α -tub 0 5 10 3 IOD (x10 ) Figure 2.2.10. Niveles de expresión de h2bs en taquizoitos (Tz) y bradizoitos (Bz). A la izquierda se observan las bandas amplificadas por RT-PCR semicuantitativa de los genes h2bv, h2ba y α-tubulina (αtub) separadas en gel de agarosa 1%. A la derecha se encuentran graficadas las intensidades de las bandas, dadas por la densidad óptica integrada (IOD) de las mismas. Estos obtenidos de tres 15 resultados experimentos independientes. Por lo tanto, podríamos decir que la expresión de h2ba se encuentra regulada a nivel del ciclo celular, mientras que la de h2bv es independiente del ciclo celular. De forma similar, en P. falciparum se observó que la expresión de PfH2B acompaña al ciclo celular, mientras que la expresión de PfH2Bv, al igual que H2Bv de Toxoplasma, es independiente del ciclo celular; probablemente indicando una función particular de esta histona variante (Miao et al. 2006) 43 )#$ 2. 3. Discusión Se identificaron tres H2Bs al realizar una búsqueda en la base de datos ToxoDB (http://toxodb.org): H2Ba, H2Bb y H2Bv. El estudio filogenético mostró dos linajes de H2B en el phylum Apicomplexa, uno que comprende a las histonas H2B variantes y otro a las canónicas. Las secuencias de las H2B del grupo de las variantes muestra mayor homología entre ellas que las del grupo de las canónicas. Estos datos sugieren que el evento de duplicación de las H2B ocurrió previamente a la divergencia del phylum Apicomplexa. Además, las H2B variantes parecen ser más conservadas que las H2B canónicas, indicando diferentes tasas de evolución de estos genes. Probablemente, los genes H2B han sufrido diferentes cambios adaptativos de acuerdo con su rol y la importancia de esta histona en el ciclo celular y la regulación de la cromatina en los diferentes parásitos Apicomplexas. La mayor diferencia de secuencia entre ellas se hallan en la región N-terminal, la cual es susceptible de ser modificada postraduccionalmente. Recientemente se describieron modificaciones postraduccionales para PfH2B y PfH2Bv (Trelle et al. 2009), donde la H2B variante posee múltiples acetilaciones en la región N-terminal, mientras que en la canónica solo encontraron la ubiquitinación en la región C-terminal. Todos los residuos acetilables de PfH2Bv se encuentran en TgH2Bv, por lo que se podría pensar que en Toxoplasma podría ocurrir lo mismo. En ese caso, se podría sugerir un papel específico a esta variante, quizás asociado a regiones de cromatina activa (ver capítulo 4). Las tres H2Bs fueron clonadas y secuenciadas utilizando ADNg y ADNc como templado. H2Bb no pudo ser amplificada de ADNc de taquizoito ni bradizoito, indicando que no es un gen transcripcionalmente activo en estos estadios. Mientras que H2Bv y H2Ba se expresan en ambos estadios con diferentes perfiles de expresión. Los niveles de transcripto de H2Ba son menores que los de H2Bv en taquizoitos y estos niveles disminuyen aún más en el estadio de bradizoito; indicando que es una histona canónica. En cambio, los niveles de expresión de H2Bv permanecen constantes en los dos estadios, o sea en los distintos estados replicativos, confirmando la calidad de variante de esta histona. En paralelo a nuestros estudios se encontraron variantes de H2B en P. falciparum (Miao et al. 2006) y T. brucei (Lowell et al. 2005). Cabe destacar que PfH2Bv también mantiene los niveles de transcripto en los diferentes estadios. En el caso de la H2Bv de T. brucei, esta no estaría relacionada filogenéticamente con la H2Bv de Apicomplexas. Estos fueron los primeros trabajos que describieron la presencia de una 44 )#$ histona H2B variante en la naturaleza. En los siguientes capítulos de abordará el estudio y la discusión de la presencia de H2Bv en protozoos. Por otra parte, se generó un anticuerpo anti-H2Bv (α-H2Bv), el cual resultó ser altamente específico y de muy baja reacción cruzada con H2Ba. Esto nos permitió estudiar esta histona a nivel de proteína. Tanto en taquizoitos como en bradizoitos esta histona se encuentra presente en el núcleo, excluyendo al nucléolo. α-H2Bv es capaz de detectar dos bandas por Western blot en ambos estadios. La intensidad de cada una de ellas fue igual tanto en taquizoitos como en bradizoitos. Así mismo, cuando se purificaron histonas de taquizoitos, se separaron por SDS-PAGE y/o TAU-PAGE y se analizaron por Western blot, también se detectaron dos bandas. Con el fin de identificar cada una de las histonas, se realizó una extracción ácida y las histonas purificadas fueron analizadas por MALDI-TOF y microsecuenciación. Esto reveló que las bandas detectadas por el anticuerpo corresponden a H2Bv y que ninguna de las bandas analizadas contenía H2Ba. La aparición de dos bandas probablemente se deba a dos estados postraduccionales diferentes de la misma histona. Todos estos resultados indican que H2Bv es la histona H2B mayoritaria en Toxoplasma. Esta conclusión es de carácter particular y sorprendente, ya que las histonas variantes se caracterizan por ser las histonas minoritarias. 45 Capítulo 3 # $# )#$! 3.1. Introducción La familia de histonas H2A es la que posee mayor número de variantes no alélicas y estas variantes difieren entre especies: H2AZ y H2AX, se encuentran en prácticamente todos los organismos eucariotas (Redon et al. 2002) mientras que H2ABbd y macroH2A, parecen ser específicas de vertebrados (Pehrson and Fried 1992, Chadwick and Willard 2001). Las diferentes histonas H2A contribuyen a la regulación de la transcripción, silenciamiento, segregación de cromosomas, mantenimiento de los telómeros y reparación del ADN. Las histonas H2As presentan una α-hélice en la región N-terminal y otra en la región C-terminal, además del plegamiento característico de las histonas. La cola Nterminal parece intercalarse entre las dos vueltas del ADN nucleosomal, mientras que la cola C-terminal contacta de forma extensa con el dímero H3-H4. La interacción entre las dos H2As del nucleosoma ocurre a través de sus dominios Loop1 (Malik and Henikoff 2003). La variante H2AZ es en general la histona H2A minoritaria. Sin embargo mostró ser esencial en todos los organismos eucariotas superiores, desde Tetrahymena a mamíferos (Liu and Gorovsky 1996, Faast et al. 2001), menos en levaduras, en las cuales la deleción génica solo produjo un crecimiento más lento de las mismas (Carr et al. 1994). Esta variante presenta en su región C-terminal un parche acídico, denominado docking domain (dominio de acoplamiento) que no se encuentra en las otras H2As (Suto et al. 2000) y pareciera ser el responsable de llevar a cabo su función esencial (Clarkson et al. 1999). Mediante la técnica de RNAi, se observó que es importante para la formación de heterocromatina, la correcta segregación de los cromosomas y la mitosis (Rangasamy et al. 2003). Además, es necesaria para el reclutamiento de la RNA polimerasa II (Adam et al. 2001) y se propone que tendría la capacidad de proteger a la cromatina transcripcionalmente activa del silenciamiento (Meneghini et al. 2003). La variante H2AX representa del 2 al 25% del pool de histonas H2As en mamíferos. Se caracteriza por presentar en el C-terminal la secuencia consenso SQ(E/D)Φ, donde Φ representa un aminoácido hidrofóbico. La ruptura de la doble hebra del ADN (DSB, del inglés double strands break) estimula la rápida fosforilación de la serina del motivo por la quinasa ATM (Rogakou et al. 1998); aunque también puede ser fosforilada por ATR y DNA-PK (Stiff et al. 2004). La exposición de las células a distintos fuentes de radiación ionizante, rayos X, radiación γ, partículas Į y metales pesados directamente inducen DSB. Los tratamientos con agentes citotóxicos como inhibidores de 47 )#$! la síntesis de ADN, agentes alquilantes del ADN, inhibidores de las topoisomerasas I y II, bleomicina y peróxido de hidrógeno, también llevan a la formación de DSB y la formación de H2AX fosforilada (γH2AX) (Dickey et al. 2009). Independientemente de cuál sea el origen, en los sitios donde hay DSB, se encuentra γH2AX. Además de participar en la reparación del ADN, γH2AX contribuye con el control del arresto celular luego de que ocurre el daño. Hasta que éste no sea reparado, el ciclo celular no progresa (FernandezCapetillo et al. 2002). Además del daño generado por agentes externos, γH2AX se encuentra asociada a los procesos celulares normales que involucran DSB, como la recombinación meiótica, la apoptosis, los rearreglos V(D)J y formación de la horquilla de replicación (Chen et al. 2000, Rogakou et al. 2000, Mahadevaiah et al. 2001, Ward and Chen 2001). La obtención y el estudio de ratones y células madres mutantes nulas para H2AX, permitió determinar que esta histona variante no es esencial para el arresto celular luego del daño del ADN, ni para la fidelidad de la recombinación V(D)J. Sin embargo, los ratones H2AX -/- presentaron retardo en el crecimiento, inmunodeficiencias, gran sensibilidad a la radiación y los machos fueron infértiles. Esto estaría indicando que esta histona es esencial en la reparación del ADN y la estabilidad genómica en mamíferos (Bassing et al. 2002, Celeste et al. 2002). Como se puede observar, las variantes de las histonas H2A son responsables del correcto funcionamiento de diversos procesos celulares. Hasta el momento de realizado este trabajo, no se habían caracterizado o estudiado las histonas de la familia H2A en Toxoplasma, como tampoco en ningún Apicomplexa. Por este motivo nos propusimos identificar las histonas H2A de T .gondii, describir similitudes y diferencias entre ellas, identificar la presencia de variantes y analizar su expresión en los estadios de taquizoito y bradizoito. 48 )#$! 3.2. Resultados 3. 2. 1. Identificación, clonado y análisis de secuencia Haciendo uso de la información y programas presentes en las bases de datos del National Center for Biotechnology Information (NCBI: http://www.ncbi.nlm.nih.gov/) y de la base de datos de T. gondii (ToxoDB: http://toxodb.org), se identificaron 3 histonas H2A putativas en este parásito. Estas fueron denominadas H2A1, H2AX y H2AZ en base la homología que presentaban sus secuencias con las ya identificadas H2As de humanos. Se clonaron mediante la técnica de PCR utilizando ADNc y ADNg como templado. La CDS de H2A1 codifica para una proteína de 128 aa con una masa molecular predicha de 13,6 kDa y punto isoeléctrico pI= 10,63 (AN: AY573602). La CDS de H2AX codifica para una proteína de 135 aa con masa molecular predicha de 14,5 kDa y pI=10,58 (AN: AY631392). Mientras que la CDS de H2AZ codifica para una proteína de 155 aa con una masa molecular predicha de 15,91 kDa y pI=10,64 (AN: AF502246). Mediante la utilización del programa ToxoDB Genome Browser (http://toxodb.org) se pudo observar que las tres cepas de parásitos secuenciadas, tipo I (GT1), tipo II (Me49) y tipo III (VEG), poseen las tres histonas H2A y éstas se encuentran en sintenia. H2A1 y H2AX muestran gran homología entre ellas (88% de identidad y 92% de similitud) y ambas se localizan en el cromosoma VIIb (H2A1: TGME49_061250; TGGT1_chrVIIb entre las posiciones 1811408 a 1811794 y TGVEG_chrVIIb entre las posiciones 18003841800743; H2AX: TGME49_061580, TGGT1_008650 y TGVEG_073130). H2AZ presenta mayor divergencia que las otras dos H2As, se encuentra localizada en el cromosoma XII y es la única que presenta intrones (TGME49_100200, TGGT1_095100 y TGVEG_007580). En la Figura 3.2.1 se muestra un alineamiento de estas tres histonas H2A. Todas tienen conservada la lisina 120, susceptible de ser ubiquitinada en otras especies y presentan las α-hélices en la región N- y C-terminal características de la familia de las H2A. Se puede observar que H2AX presenta el motivo C-terminal característico SQ(E/D)Ɏ, que en este caso es SQEF. Resulta interesante que H2A1 posee una versión truncada del motivo que define a la H2AX, por lo que no se puede descartar que esta histona posea funciones que se han visto asociadas a H2AX. H2AZ presenta una región N-terminal larga, como se ha observado en otros protozoos (Sullivan et al. 2006) y presenta varias diferencias con respecto a las otras H2As de T. gondii; la mayoría de las cuales se encuentran en las regiones N- y C- terminales. 49 )#$! 1 ------------------MSAKGKGGRAKKS-GKSS----SKSAKAGLQFPVGRIGRYLKKGRYAK-RVGAGAPVYMAAV 1 ------------------MSAKGAGGRKKTSSGKKV----SRSAKAGLQFPVSRIGRYLKKGRYAK-RVGVGAPVYLAAV 1 MDGAGKVGGKVGGKVGGKVGGMGKGGKGKSGSGKGKKAPLSRAARAGLQFPVGRVHRMLKSRISSEGRVGSTAAVYASAI L1 αΝ H2A1 H2AX H2AZ L2 α1 α2 Ub 59 LEYLCAEILELAGNAARDHKKTRIIPRHIQLAVRNDEELSKFLGGVTIASGGVMPNVHSVLLPK--KSKGKK--SQ 59 LEYLCAEILELAGNAARDHKKTRIIPRHIQLAVRNDEELSKFLGGVTIANGGVMPHVHAVLLPKHSKSKGKHGVSQEF 81 LEYLTAEVLELAGNASKDLKVKRITPRHLQLAIRGDEELDTLIK-ATIAGGGVIPHIHKSLMTKGPSTQPMKKAKK L3 α2 H2A1 H2AX H2AZ L4 α3 αC Figura 3.2.1. Alineamiento de las H2As de T. gondii. Alineamiento de la secuencia de aminoácidos de H2A1, H2AX y H2AZ. El sitio predicho de ubiquitinación (Ub) se encuentra sombreado en negro. Debajo de la secuencia se encuentra una representación esquemática de la estructura secundaria donde α y L significan α-hélice y loop, respectivamente; creada utilizando el programa Predator (VegaZZ v2.3). La presencia de Polimorfismo de un solo nucleótido (SNPs) para cada una de estas histonas en cepas de los tres tipos, se obtuvo de ToxoDB. H2AZ presenta gran número de SNPs, sin embargo todos ellos se encuentran en regiones no codificantes; mostrando una densidad nula de SNPs en la región codificante (Tabla 3.2.1). Por el contrario, H2AX y H2A1 poseen baja densidad de SNPs (1 y 3 respectivamente) en la región codificante; no obstante todos los cambios son sinónimos (Tabla 2.2.1). Esto indica una selección purificadora, correlacionada con la importancia de conservar estas proteínas. Densidad SNPs SNPs en Totales CDS No sinónimos No Sinónimos codificante s Gen Cepas Comparadas H2A.Z 145.m00002 ME49 vs VEG GT1 vs ME49 ME49 vs RH 0 0 0 60 55 5 0 0 0 0 0 0 60 55 5 H2A.X 55.m04942 GT1 vs VEG GT1 vs ME49 2,45 2,45 1 1 0 0 1 1 0 0 H2A.1 55.m04926 ME49 vs VEG GT1 vs ME49 GT1 vs VEG 7,75 7,75 0 6 5 3 0 0 0 3 3 0 3 2 3 En concordancia con estos resultados, el análisis de Neighbor Joining muestra que la H2AZ de T. gondii se agrupa con H2AZ de otros organismos antes que con las otras H2As del parásito. Esto estaría indicando, como era de esperar, un origen evolutivo único y anterior a la diversificación de las especies. Por otro lado, H2A1 y H2AX se 50 )#$! agrupan en un clado que contiene a las H2A canónicas y H2AX de otras especies (Figura 3.2.2 y (Sullivan et al. 2006)). HuH2A 88 99 XlH2A 96 HuH2AX 96 DmH2A ScH2A1 64 100 ScH2A2 AtH2A TgH2A1 86 51 TgH2A2 TgH2AX 57 PfH2A 99 100 PyyH2A TtH2AZ AtH2A.F/Z 100 PfH2AZ 96 100 93 PyyH2AZ TgH2AF/Z XlH2A.Z 56 HuH2AZ 99 88 DmH2AZ TtH2A1 100 TtH2A2 GlH2A 100 GlH2AX Figura 3.2.2. Arbol de Neighbor joining (NJ). Se realizó con la secuencia de aminoácidos de las H2As de los siguientes organismos. En el recuadro se encuentra la subfamilia de las H2AZ. At, Arabidopsis thaliana y (ANs: AAL85051 BAF00012), Dm, Drosophila melanogaster (ANs: AAN11125 y AAF56631), Gl, Giardia lamblia (AN: AF139873); Hu, human (ANs: AAN59958, P16104 y Pf, CAG33696), Plasmodium falciparum (ANs: P40282 y CAB39069); Pyy, Plasmodium yoelii yoelii (ANs: CAQ40903 y Sc, EAA15833); Saccharomyces cerevisiae (ANs: P04909, P04910 y Q12692), Tg, Toxoplasma gondii, Tt, Tetrahymena thermophilus (ANs: AAC37292, AAC37291 y CAA33554), Xl, Xenopus laevis (ANs: AAA49769, AAH74188 y AAH74203). 0.1 Las histonas H2A de Toxoplasma presentan un alto grado de divergencia con respecto a otros organismos, a diferencia de las histonas H3 y H4, las cuales se encuentran altamente conservadas en la mayoría de los eucariotas (Sullivan 2003, Sullivan et al. 2006). T. gondii H3 y H4 presentan un 94-98% de homología con las H3 y H4 de humanos y S. cerevisiae. De modo contrario, TgH2AX muestra solo un 78-83% de similitud (~69% de identidad) y TgH2AZ un 80-98% de similitud (67-81% de identidad) con sus homólogas en humanos y S. cerevisiae, respectivamente. A su vez, TgH2A1 comparte un 86% de similitud y un 73% de identidad con la H2A canónica de humanos. Si bien las modificaciones postraduccionales de las H2As no han sido ampliamente estudiadas, se supone que ocurren al igual que en el resto de las histonas. En la Figura 3.2.3 se muestra un alineamiento entre las regiones N-terminales de las tres histonas H2A de Toxoplasma y las modificaciones postraduccionales descriptas en S. 51 )#$! cerevisiae (ScH2B) y humanos (HuH2B). Las H2As de Toxoplasma poseen varias lisinas, argininas y serinas que podrían ser potencialmente acetiladas, metiladas y/o fosforiladas. La serina en posición dos (S2) capaz de ser fosforilada en la H2A1 de humanos, se encuentra conservada en TgH2A1, al igual que K6. No obstante, resulta difícil deducir los residuos de las H2As de Toxoplasma que estarían modificados, debido a la divergencia existente entre las secuencias, sobre todo en la variante H2AZ (Figura 3.2.3). Recientemente se describieron las modificaciones postraduccionales para las histonas H2A de P. falciparum (PfH2A1 y PfH2AZ) (Trelle et al. 2009), observando que PfH2AZ posee múltiples acetilaciones en su región N-terminal, mientras que en la canónica se encontraron solo dos lisinas acetiladas (K4 y K6). Si bien en humanos la H2AZ presenta mayor número de acetilaciones que la canónica, el número total de residuos acetilados es mucho menor que el observado en Plasmodium. Todos los residuos acetilados en P. falciparum se encuentran conservados en TgH2A1 y TgH2AZ (Figura 2.2.2), por lo que se podría pensar que en Toxoplasma estaría ocurriendo lo mismo. En un trabajo reciente en S. cerevisiae (Parra and Wyrick 2007), se observó que la región N-terminal de H2A es responsable de la represión de aproximadamente el 4,5% de su genoma y que el principal responsable es un motivo que denominaron dominio HAR (dominio represor de H2A, del inglés histone A represive) y comprende la secuencia “QSRSA”. La búsqueda de este motivo en las histonas H2As de Toxoplasma reveló que TgH2AX presenta este motivo con mayor grado de conservación (Figura 3.2.3). MSAKGKGGRAKKSGKSSSK MSAKGKTGR-KKASKGTSN MSGRGKQGG-KARAKAKSR SAKAGLQFPVGRI SAKAGLQFPVGRI SSRAGLQFPVGRV TgH2A1 PfH2A1 HuH2A1 MSAKGAGGRKKTSSGKKVSR SAKAGLQFPVSRI MSGRGKTG--GKARAKAKSR SSRAGLQFPVGRV MSG-GKSGGKAAVAKSAQSR SAKAGLAFPVGRV MSG-GKGGKAGSAAKASQSR SAKAGLTFPVGRV Dominio HAR TgH2AX HuH2AX ScHTA1 ScHTA2 M Fosforilación M Acetilación MDGAGK-VGGKVGGKVGGKVGGMGKGGKGKSGSGKGKKAPLSR MEVPGKVIGGKVGGKVGGKVLGLGKGGKGKTGSGKTKKAPLSR ----------MAGGKAG-------------KDSGKAKTKAVSR -------MSGKAHGGKGKS---------GAKDSGSLRS--QSS AARAGLQFPVGRV ASRAGLQFPVGRV SQRAGLQFPVGRI SARAGLQFPVGRI TgH2AZ PfH2AZ HuH2AZ ScHTZ1 Figura 3.2.3. Comparación de la secuencia N-terminal de las H2A. Tg: T. gondii histones. Pf: P. falciparum histones Hu: human histones (ANs: AAN59958, P16104 y CAG33696). Sc: S. cerevisiae (AN: P04909, P04910 y Q12692). HTA1 y HTA2 son histonas H2AX y HTAZ1 es H2AZ. El panel muestra las modificaciones postraduccionales identificadas en las H2As de humanos, levaduras y Plasmodium. Los residuos modificados se encuentran sombreados y el color indica el tipo de modificiación de acuerdo a lo indicado en el cuadro. El dominio represor de H2A se encuentra subrayado (dominio HAR). El cilindro indica la α-hélice N-terminal de las H2As. 52 )#$! 3. 2. 2. Obtención de anticuerpos específicos Las tres H2As de T. gondii fueron expresadas como proteínas recombinantes: rH2AZ, rH2AX and rH2A1. Todas fueron subclonadas en vectores de expresión en bacterias, expresadas mediante la inducción con IPTG y purificadas en columnas de niquel como se describe en el capítulo 6. Las tres proteínas recombinantes migraron con diferentes masas moleculares aparentes al ser analizadas por SDS-PAGE 15%. Estas se encuentran entre 18 y 20 kDa (Figura 3.2.4). Además, los 120 aa N-terminales de la histona H2AZ se expresaron como proteína recombinante fusionados a la proteína glutation Tio-Transferasa [GST] (denominada GST-Nt). Esta proteína recombinante migró con una masa molecular aparente de aproximadamente 30 kDa (Figura 3.2.4), mayor que la observada para GST sola (dato no mostrado). Figura 3.2.4. Histonas H2A recombinantes. Las secuencias codificantes de H2AX y H2A1 fueron subclonadas en el plásmido pRSETA (Invitrogen), mientras que la de H2AZ en pQE30 (Quiagen). Todas fueron expresadas como proteínas recombinantes, las cuales contienen una cola de polihistidinas que permitió su purificación mediante una columna de niquel: rH2A1, rH2AX y rH2AZ. Por otro lado, se expresó la región N-terminal de H2AZ, la cual fue expresada como proteína recombinante fusionada a GST (GST-Nt). En la figura se observan las proteínas purificadas separadas en SDS-PAGE 15% y teñidas con Coomassei blue. Con el fin de obtener anticuerpos específicos para cada una de estas histonas, rH2A1 y GST-Nt fueron utilizadas para inmunizar ratones, mientras que rH2AX y rH2AZ se emplearon para inmunizar conejos. Se obtuvieron sueros policlonales específicos para cada una de ellas, lo cual fue analizado por Western blot utilizando las proteínas recombinantes. De los ratones inmunizados con rH2A1 se obtuvieron dos lotes de anticuerpo: uno altamente específico α-H2A1 L1 y otro que presenta reacción cruzada con rH2AX: α-H2A1 L2. El antisuero α-H2AX reconoce de forma específica rH2AX y es no reactivo frente a rH2A1 ni rH2AZ. Tanto α-H2AZ de conejo, como el antisuero de ratón α-H2AZ-Nt, son altamente específicos para rH2AZ y no reconocen rH2A1 ni rH2AX. 53 )#$! Figura 3.2.5. Estudio de la especificidad de los anticuerpos α-H2As por Western blot. En todos los casos 1,5 μg de cada proteína recombinante (rH2A1, rH2AX y rH2AZ) fueron separadas por SDS-PAGE15%, transferidas a membrana de nitrocelulosa y teñidas con Ponceau (fotos en rosa). Esto permite observar las bandas de cada histona recombinante presente. Luego se realizaron Western blots con los anticuerpos especificados a la izquierda de cada panel. Además, se determinó la afinidad de cada anticuerpo por cada H2A recombinante mediante ELISA competitivo. α-H2AX pierde la capacidad de unirse a rH2AX inmovilizada cuando es preincubado con esta proteína recombinante. Mientras que la preincubación del anticuerpo con rH2AZ, rH2A1 o rROP2 no afecta la interacción entre α-H2AX y la rH2AX inmovilizada, incluso en concentraciones de 10 μg/ml (Figura 3.2.6). De la misma manera, la unión entre α-H2AZ y rH2AZ inmovilizada solo se ve afectada cuando este anticuerpo es preincubado con la misma proteína soluble. En concordancia con lo observado por Western blot, la unión de α-H2A1 L2 a rH2A1 inmovilizada se ve disminuida al ser preincubado con rH2A1 y rH2AX; aunque rH2AX afecta menos que rH2A1. La afinidad de α-H2A1 L1 no pudo ser evaluada por no contar con suficiente cantidad de anticuerpo (Figura 3.2.6). 54 )#$! Figura 3.2.6. Afinidad de los anticuerpos anti-H2As determinada por ELISA de competencia. Cada panel corresponde al análisis de la afinidad de un anticuerpo, indicado como título del mismo, con respecto a la proteína recombinante específica inmobilizada: αH2A1 L2 con respecto a rH2A1; α-H2AX con respecto a rH2AX y α-H2AZ con respecto a rH2AZ. En todos los casos cada anticuerpo fue preincubado con distintas concentraciones de rH2A1, rH2AX, rH2AZ y rROP2, cada una indicada con el símbolo y línea indicado en el recuadro en la parte superior derecha. 3. 2. 3. Inmunodetección de las histonas de la familia H2A Una vez demostrada la especificidad de los anticuerpos para cada una de las H2As, se procedió a identificar cada una de las histonas endógenas del parásito. Se realizó un ensayo de Western blot con extracto total de taquizoitos separados por NuPAGE 4-12% (Invitrogen). Cada anticuerpo reconoce una única banda; todas con una masa molecular aparente similar (Figura 3.2.7A). Por lo tanto, se realizó una purificación de histonas de taquizoitos, las cuales fueron separadas por SDS-PAGE 15%, transferidas a una membrana de nitrocelulosa y visualizadas mediante tinción con rojo Ponceau. Luego, cada una de las calles fueron divididas a la mitad y analizadas por Western blot con dos anticuerpos diferentes. De este modo es posible visualizar diferencias mínimas en la masa molecular aparente de las mismas. Se compararon las bandas detectadas 55 )#$! con α-H2AZ-Nt y α-H2A1 L1 con respecto a las bandas reconocidas por α-H2AX. De este experimento se deduce que cada histona H2A de Toxoplasma posee un masa molecular aparente diferente. Además, se puede decir que H2AZ es la minoritaria ya que la banda detectada por α-H2AZ-Nt no puede ser observada con rojo Ponceau en comparación a las bandas detectadas por α-H2AX y α-H2A1 L1. Sin embargo, no es posible determinar las cantidades relativas de estas histonas porque otras histonas (como H3) pueden comigrar con ellas (Figura 3.2.7B). Figura 3.2.7. Detección de las histonas H2As de T. gondii por Western blot (WB). A. Extracto de taquizoitos separados por NuPAGE 4-12%. B. Histonas purificadas separadas por SDS-PAGE 15%. Las histonas se visualizan con rojo Ponceau. A su vez, se determinó por inmunofluorescencia indirecta que cada una de estas histonas se encuentra en el núcleo de los taquizoitos, exceptuando el nucléolo (Figura 3.2.8). 56 )#$! Figura 3.2.8. Localización subcelular de las H2As. Se llevó a cabo mediante inmunofluorescencia indirecta en taquizoitos con cada uno de los anticuerpos (indicados a la izquierda del panel). A su vez, fueron teñidas con DAPI, el cual se intercala en el ADN marcando asi los núcleos. Luego se realizó una superposición de ambas señales para ver si hay colocalización. DAPI también tiñe el núcleo de las células huesped (Hu). Las flechas apuntan a los nucleolos. 3. 2. 4. Perfiles de expresión de las histonas de la familia H2A Los perfiles de expresión de los genes que codifican las H2As fueron estudiados en taquizoitos y bradizoitos in vitro. Los taquizoitos RHΔuprt intracelulares fueron incubados por un lado en condiciones de taquizoitos y por otro lado a pH 8,1 y sin CO2. La aparición de bradizoitos en este último caso fue analizado por inmunofluorescencia indirecta observando la aparición de BAG1 y la pared del quiste con la lectina de Dolichos biflorus fluorescente (Figura 3.2.9A). También se determinó el aumento de los transcriptos correspondientes a bag1 y ldh2 (genes específicos de bradizoitos) y la disminución de sag1 (específico de taquizoito) por PCR en tiempo real en un análisis comparativo (qRT-PCR). Los bradizoitos presentan un aumento significativo de los niveles de transcripto de h2aX (p<0,0001) y de h2a1 (p ޒ0,05) con respecto a sus niveles en taquizoitos. A diferencia de los niveles de h2aZ, los cuales permanecen constantes en ambos estadios (Figura 3.2.9B). En todos los casos, los niveles de cada gen fueron normalizados con respecto a β-tubulina (que no presenta cambios significativos en bradizoitos in vitro (Weiss et al. 1998)). 57 )#$! Figura 3.2.10. Expresión de las H2As en taquizoitos (Tz) y bradizoitos (Bz) in vitro. A. La eficiencia de diferenciación (mayor al 80%) fue determinada por inmunofluorescencia indirecta realizada con LDB y BAG1. B y C. Se extrajo ARNm de éstos parásitos y de taquizoitos, los cuales fueron utilizados como templado en una cuantificación relativa por PCR en tiempo real. Los niveles de transcriptos de cada gen fueron normalizados con respecto a β-tubulina y calibrados con respecto a taquizoitos. Los niveles de genes específicos de bradizoitos (bag1 y ldh2) y taquizoitos (sag1) con el fin de confirmar la diferenciación (B) y de cada una de las H2As (C). Se realizaron tres experimentos independientes. Las diferencias significativas entre Tz y Bz fueron analizados por test T de Student (GraphPad Prism software): * indica p<0,05 y *** indica p<0,001. En B todos presentan una pޒ0,001. Se graficaron como logaritmo en base 2 de las veces que cambia la expresión de cada gen en Bz con respecto a Tz. Estos resultados aumentan las posibilidades de que h2aX y h2a1 estén involucrados en la diferenciación a bradizoitos, asociados a un estado específico del ciclo celular o ambos. Como los bradizoitos maduros obtenidos in vivo se encuentran arrestados en G0 o G1 con un contenido uniforme de ADN correspondiente a 1N (Radke and White 1998, Radke et al. 2003), se determinaron los perfiles de expresión de los genes de las histonas H2As en bradizoitos de quistes aislados de cerebro de ratón. Para ello se realizaron PCRs semicuantitativas utilizando ARNm de quistes extraídos de ratones, 30 días después de su infección. La figura 3.2.11 muestra que h2a1 no se expresa en bradizoitos maduros, o lo hace en cantidades indetectables por este método, mientras que si lo hacen h2aX y h2aZ. Como las histonas canónicas se expresan exclusivamente durante la fase S del ciclo celular (Harris et al. 1991), se puede deducir que H2A1 es, por lo tanto, la H2A canónica en Toxoplasma. 58 )#$! Tz Quiste de cerebro (Q) h2a1 h2aX h2aZ β -tub Q Figura 3.2.11. Expresión de H2As en taquizoitos (Tz) y bradizoitos de quistes de cerebro de ratón (Q). Se extrajeron quistes de cerebros de ratón 30 días después de su infección, se contaron y visualizaron por microscopía de contraste de fases. Se les extrajo ARN, el cual fue utilizado como templado para analizar la expresión de las H2As por PCR semicuantitativa. Como control de carga se amplificó el gen de β-tubulina (β-tub). 3. 2. 5. H2AX y daño del ADN en taquizoitos La variante H2AX se fosforila rápidamente en su motivo C-terminal SQ(E/D)φ luego de la ruptura de la doble hebra del ADN (Downs et al. 2000). Se han descripto numerosos agentes capaces de producir diferentes tipos de daños del ADN e inducir la fosforilación de esta histona (revisados por (Dickey et al. 2009)). Con el fin de dilucidar si este mecanismo se encuentra conservado en Toxoplasma, se analizó el efecto de un agente oxidante (H2O2) y uno alquilante (metano sulfonato de metilo [MMS]) sobre la fosforilación y expresión de H2AX en taquizoitos. En un primer experimento se incubaron parásitos extracelulares con 0, 100, 200 y 400 μM de H2O2 por 1 hora a 37°C, se lisaron y analizaron por NuPAGE 4-12% seguido de Western blot. Se utilizó un anticuerpo monoclonal de ratón capaz de reconocer el motivo SQEY fosforilado en la serina, denominado α-γH2AX y el anticuerpo α-H2AX generado en el laboratorio. Ambos anticuerpos reconocen una sola banda con la misma migración bajo las condiciones descriptas. Como control de la cantidad de proteína presente se utilizó anti-tubulina (α-Tub). Se cuantificaron las intensidades de las bandas y se determinaron las cantidades relativas de cada histona con respecto a Tub (R). De este modo es fácil ver que los niveles de H2AX permanecen constantes, mientras que los de γH2AX aumentan frente al tratamiento con H2O2 de manera dependiente de la dosis (Figura 3.2.12). Si bien se sabe que las variantes de histonas presentan una transcripción independiente del ciclo celular (Grove and Zweidler 1984), hasta el momento no se han descripto mecanismos que regulen su expresión. Como el tratamiento con H2O2 induce la fosforilación de H2AX, este agente podría estar afectando de algún modo su transcripción. Por lo tanto, se determinaron las expresiones de las tres H2As por qRTPCR en parásitos 59 )#$! tratados del mismo modo antes descriptos. Los resultados muestran que la transcripción de h2a1 y h2aX es estimulada por H2O2 de manera dosis-dependiente, pero no afecta a h2aZ (Figura 3.2.12). Es posible observar que frente al estrés oxidativo, luego de media hora, se observa un aumento en los niveles de transcripto de H2AX, el cual no se ve reflejado en un aumento en los niveles de proteína, pero si en un aumento en la fosforilación de la misma. Es probable que el tiempo no sea suficiente para que el aumento en los niveles de ARNm se correlacionen con los de proteína. A H2O2 (μ μ M) B 0 100 200 400 α -γγ H2AX R H2O2 (μ μ M) 0 100 200 400 α -Tub α -H2AX 6 Aumento (veces) α -Tub H2O2 5 4 3 2 1 0 0 100 200 400 0 100 200 400 0 100 200 400 μ M h2a1 h2a1 h2aX h2aX h2aZ h2aZ R Figura 3.2.12. Efecto de daño oxidativo con H2O2 en taquizoitos. Taquizoitos extracelulares fueron tratados con 0, 100, 200 y 400 μM de H2O2 por 1 h a 37 °C. A. Western blots realizados con anti-H2AX en el panel inferior y anti-fosfoH2AX (α-γH2AX) en el panel superior. Ambos fueron realizados también con anti-tubulina (α-Tub), utilizado como control interno. La intensidad de las bandas fue cuantificada y se estableció la relación de la intensidad de las histonas con respecto a tubulina (R). B. Análisis de la expresión de h2a1, h2aX y h2aZ por qRT-PCR normalizada con βtubulina y calibrado con respecto al control sin H2O2. El ráfico representa uno de tres experimentos independientes. Los mismos experimentos fueron llevados a cabo con MMS. Ningún cambio fue observado en los niveles de H2AX, ni en la fosforilación de la misma, ni en los niveles de transcripto de ninguna de las H2As, incluso a concentraciones mayores, como 800 μM (Figura 3.2.13). Tampoco se observaron cambios en estos parámetros cuando se trataron los parásitos con concentraciones de MMS de 50 y 100 μM (datos no mostrados). 60 )#$! MMS (μ μ M) 0 200 400 800 α -Tub α -γγ H2AX MMS (μ μ M) 0 200 400 800 α -Tub B MMS 6 Aumento (veces) A 5 4 3 2 1 0 0 200 400 800 0 200 400 800 0 200 400 800 μ M h2a1 α -H2AX H2A.1 h2aX H2A.X h2aZ H2A.Z Figura 3.2.13. Efecto de daño alquilante con MMS en taquizoitos. Taquizoitos extracelulares fueron tratados con 0, 200, 400 y 800 μM de MMS por 1 h a 37 °C. A. Western blots realizados con anti-H2AX en el panel inferior y anti-fosfoH2AX (α-γH2AX) en el panel superior. Ambos fueron realizados también con anti-tubulina (α-Tub), utilizado como control interno. B. Análisis de la expresión de h2a1, h2aX y h2aZ por qRT-PCR normalizada con β-tubulina y calibrado con respecto al control sin H2O2. El ráfico representa uno de tres experimentos independientes. Cabe destacar que los taquizoitos sometidos a estrés térmico, el cual consiste en 2 horas a 42 °C, no presentan ningún aumento en los niveles de transcripto de h2aX (Figura 3.2.14). Aumento (veces) H2AX 1,5 Figura 3.2.14. Expresión de h2aX en estrés térmico. Se colectaron taquizoitos extracelulares; la mitad fueron incubados a 42 °C por 2 h, mientras que la otra mitad fue incubada a 37 °C como control. Se les extrajo ARN y se determinaron los niveles de h2aX por qRT-PCR. Los valores se encuentran normalizados con β-tubulina y calibrados con respecto al control de 37 °C. 1 0,5 0 37C 42C Estos datos sugieren que la sobreexpresión de h2aX y h2a1, como el incremento en la fosforilación de H2AX, se encuentran asociadas al daño oxidativo del ADN y/o celular. 61 )#$! 3.2.6. La sobreexpresión de H2AX y su papel en la replicación del parásito Con el fin de intentar establecer la relación existente entre el aumento de la expresión de H2AX y diferentes procesos biológicos, se obtuvieron parásitos RH que sobreexpresan esta histona de forma estable, fusionada a c-myc en la región N-terminal (denominados RH mycH2AX). Se aislaron tres clones: C2, C3 y C4, los cuales fueron analizados por Western blot. α-H2AX reconoce una sola banda en los parásitos salvajes y dos bandas en los clones, siendo la banda extra de mayor masa molecular aparente. Mientras que α-c-myc reconoce una banda sólo en los clones, cuya masa molecular aparente coincide con la banda extra observada con α-H2AX (Figura 3.2.15A). Estos datos confirman la expresión de mycH2AX recombinante en los tres clones. La intensidad de las bandas reconocidas con α-H2AX (H2AX endógena + mycH2AX) fueron cuantificadas utilizando el programa Gel Pro Analyzer v. 4.0, en relación con la intensidad de las bandas detectadas con α-Tub. Todos los clones presentan casi dos veces más niveles de H2AX que los parásitos salvajes (Figura 3.2.15B). Figura 3.2.15. Análisis de los clones que sobreexpresan H2AX. A. Western blot con α-tubulina (α-Tub), α-H2AX y α-c-myc de taquizoitos de los clones RH mycH2AX C1, C2 y C3 y de la cepa salvaje (WT). B. Gráfico de la intensidad de las bandas detectadas con αH2AX en cada línea celular con respecto a la detectada en WT. IOD: densidad óptica integrada Además, se realizó una inmunofluorescencia indirecta con α-c-myc en taquizoitos intracelulares de los tres clones RH mycH2AX, detectándose señal en los núcleos. Confirmando de este modo la correcta localización de mycH2AX (Figura 3.2.16). 62 )#$! Figura 3.2.16. Inmunofluorescencia indirecta (IFI) de RH mycH2AX. Cada uno de los clones (C2, C3 y C4) fue analizado con α-c-myc por IFI. Los núcleos fueron teñidos con DAPI. Luego se realizó una superposición de estas dos imágenes corroborando una colocalización de la señal. Una vez seleccionados tres clones que sobreexpresan H2AX, la cual presenta la misma localización que la proteína endógena, se realizó un ensayo preliminar de replicación con el objetivo de estudiar posibles cambios en el fenotipo. Se comparó el crecimiento de los clones con respecto a los parásitos salvajes (WT). Los taquizoitos extracelulares de cada cepa fueron contados, lavados e incubados con células HFF en frío por 1 h para estimular su invasión. Luego se lavaron todos los parásitos extracelulares y se incubaron en condiciones de taquizoito o.n. (ver capitulo 6). Al día siguiente se fijaron los cultivos y se contaron parásitos por vacuola en cada caso. Se observó que los clones 2 y 3 crecieron más con respecto al clon 4 y el WT, de modo significativo con Pޒ0,01; mientras que el crecimiento del clon 4 no fue significativamente diferente con respecto a la cepa salvaje (Figura 3.2.17). Es importante destacar que este experimento fue realizado por única vez, por lo que son resultados preliminares. Figura 3.2.17. Análisis de replicación. Se comparó la velocidad de replicación de los clones (C2, C3 y C4) con respecto a la cepa salvaje (WT). Para cada uno se detalla el porcentaje de vacuolas parasitóforas (%VP) conteniendo 1, 2, 4 y 8 taquizoitos (Tz/VP). Los clones 2 y 3 crecieron más que C4 y WT significativamente con Pޒ0,01 (**). 63 )#$! 3.1. Discusión Se han descripto numerosas variantes de H2A: H2AX, H2AZ, macroH2A y H2ABbd. Las últimas dos se han observado solo en vertebrados. Las histonas H2As difieren entre las distintas especies. Los protozoos Trypanosoma spp no tienen H2AX; por el contrario las levaduras, Giardia lamblia, Entamoeba histolytica y Trichomonas vaginalis han reemplazado la H2A canónica por H2AX; mientras que Drosophila melanogaster presenta una variante de H2A (H2AvD), la cual es un híbrido entre H2AX y H2AZ (van Daal et al. 1988). Al estudiar lo que ocurre en Apicomplexas, se observó que Toxoplasma es el único que posee H2A canónica y H2AX. Cryptosporidium spp., al igual que los protistas mencionados anteriormente, parecen haber remplazado la histona H2A por H2AX. Los otros Apicomplexas analizados no tienen H2AX; sin embargo, sus H2A canónicas presentan homología parcial con el motivo C-terminal SQ(E/D)Ɏ característico de las H2AX (Sullivan et al. 2006). Por lo que no se puede descartar que estas H2As estén cumpliendo el rol de H2AX. Por el contrario, todos los Apicomplexas tienen H2AZ y presentan una gran homología entre si. H2AZ de T. gondii presenta una región N-terminal de mayor longitud, con un mayor número de K, que HuH2AZ y ScH2AZ. En P. falciparum, H2AZ también presenta una región N-terminal larga y se encuentra hiperacetilada (Trelle et al. 2009). Todos los residuos modificados de PfH2AZ se encuentran conservados en TgH2AZ (Figura 3.2.3), lo cual sugiere que lo mismo podría ocurrir en Toxoplasma. El análisis filogenético de las histonas H2As revela que H2AZ de T. gondii se agrupa con las H2AZ de otros organismos y no con las otras H2As de Toxoplasma. Esto indica que esta histona presenta un origen evolutivo único, como era de esperar (Malik and Henikoff 2003), que siempre se mantuvo diferente al de las otras H2As. Por otro lado, las histonas H2A1 y H2AX de T. gondii se agrupan junto con las H2A canónicas y H2AX de otros Apicomplexas. La presencia de H2A1 y H2AX varían mucho en los diversos organismos y se propone que H2AX podría presentar múltiples orígenes evolutivos (Malik and Henikoff 2003, Li et al. 2005a), aunque también se piensa que las histonas canónicas podrían haber surgido de la variante H2AX. Los datos de secuencia obtenidos en Apicomplexas, incluyendo el motivo AQEF (en vez de SQEF) de Plasmodium spp. (Sullivan et al. 2006), sugiere esta última opción. La aparición de esta variante podría estar relacionada con alguna característica particular de la especie. La principal función de esta histona es mantener la estabilidad genómica. Plasmodium ssp., Trypanosoma ssp. y Leishmania ssp. no tienen H2AX y se ha descripto que presentan una gran plasticidad genómica, en los cuales los tamaños de 64 )#$! ciertos cromosomas varían entre diferentes cepas (Hernandez-Rivas 1996, Henriksson J 1995, Cruz AK 1993). P. falciparum presenta una alta fragilidad en las regiones teloméricas y subteloméricas, lo cual estaría promoviendo la variación en los genes de virulencia. Por otro lado, Toxoplasma posee H2AX y no se ha observado que presente inestabilidad genómica (Frenkel JK 1996). Se podría hipotetizar que los parásitos que tienen genomas más plásticos no poseen H2AX, mientras que los que presentan un genoma más estable si. Sería necesario un experimento de deleción génica de h2ax de T. gondii para corroborar esta hipótesis. Se obtuvieron anticuerpos específicos para cada una de las H2As, lo cual permitió la identificación selectiva de cada una de ellas, generando las herramientas fundamentales para su análisis y caracterización. El uso de los mismos en ensayos de Western blot de histonas purificadas, reveló que las tres H2As se encuentran presentes en taquizoitos y H2AZ es la minoritaria, acorde con lo observado en otros organismos (Wu and Bonner 1981). Consistentemente con lo observado a nivel de proteína, las tres H2As fueron amplificadas de ADNc de taquizoitos. Cuando se comparan los niveles de transcripto con los de bradizoitos obtenidos in vitro se observó un aumento de los niveles de h2a1 y h2aX, mientras que los niveles de h2aZ se mantienen constantes. Cuando se los compara con los niveles de transcripto de bradizoitos obtenidos in vivo, solo se observa la expresión de h2aX y h2aZ. Los parásitos inducidos a bradizoitos, se encuentran arrestados en fase S tardía-G2 del ciclo celular con un contenido de ADN de 1.8N; mientras que los bradizoitos de quistes maduros se encuentran en fase G0-G1 con un contenido de ADN de 1N (Radke et al. 2003). Como consecuencia, estos resultados indican que h2a1 se expresa en las poblaciones de parásitos que pueden entrar en la fase S del ciclo celular; lo cual indica la calidad de canónica de esta histona. Por otro lado, h2aZ y h2aX mantienen los mismos perfiles de expresión en ambos tipos de bradizoitos, confirmando su calidad de variantes. Como se mencionó arriba, la expresión de h2aX aumenta en bradizoitos in vitro (arrestados en fase S tardía-G2) e in vivo (arrestados en G0-G1). Los linfocitos que carecen de H2AX (H2AX-/-) son incapaces de generar el arresto celular en G2-M después de que se produce daño en el ADN (Fernandez-Capetillo et al. 2002). Este resultado sugiere que H2AX es necesaria para que el arresto ocurra en ese momento. Podría ser que el aumento en la expresión de h2aX en bradizoitos se deba a la necesidad de esta histona para que los parásitos se diferencien, ya que como vimos, el 85% de los parásitos que se están diferenciando se encuentran arrestados en S tardía-G2. El aumento de la expresión de h2ax en bradizoitos es completamente novedoso. Podría ser que esta 65 )#$! histona sea necesaria para la diferenciación y/o que esté jugando un papel importante en el mantenimiento del estadio de bradizoito (ver también discusión del capítulo 4). Empleando un anticuerpo comercial pudimos demostrar la presencia de γH2AX en el parásito. Si bien el análisis por Western blot sugiere que este anticuerpo está reconociendo solo a H2AX, sería interesante estudiar si el motivo C-terminal truncado (SQ) de H2A1 también es capaz de ser fosforilado. La fosforilación de la H2AX se ha descripto en varios organismos y se encuentra asociada a reparación del ADN (Downs et al. 2000). Esto también ocurre en Toxoplasma, donde la presencia de H2O2 estimula la formación de γH2AX en taquizoitos, con un aumento dependiente de la dosis. Sin embargo esta inducción no ocurre en presencia de MMS, lo cual podría deberse a la ausencia de DBS. Lundin y colaboradores (Lundin et al. 2005) mostraron fuertes evidencias de que este agente in vivo causaría fragilidad del ADN frente al calor y no DSB. Es importante destacar que los taquizoitos extracelulares no tratados presentan cierto nivel de H2AX fosforilada. O sea que γH2AX además de estar mediando la reparación del daño producido en el ADN, estaría desempeñando otra función en el estado basal de los taquizoitos. McManus et al (McManus and Hendzel 2005) propone que en las células de mamíferos existen dos grupos de γH2AX: uno involucrado en la reparación del ADN, que forma grandes foci que se localizan preferentemente en eucromatina, capaz de reclutar proteínas involucradas en la reparación. El otro, forma pequeños foci, localizados de forma similar en eucromatina y heterocromatina, que no colocaliza con ningún factor de reparación del ADN y su cantidad relativa se encuentra asociada al ciclo celular, alcanzando el pico máximo durante la mitosis. Sin embargo, la función de este aumento en la fosforilación de H2AX asociada a mitosis es desconocida. Resulta interesante que los niveles de transcripto de h2aX y en menor medida los de h2a1, aumentan en respuesta al tratamiento con H2O2, lo cual no ocurre con cualquier otro tipo de estrés como el choque térmico. Esto podría deberse a un estímulo generado por la necesidad de H2AX para la reparación del ADN, por el estado redox presente dentro de la célula, o que el daño oxidativo se encuentre estimulando la diferenciación a bradizoitos. Este es el primer reporte acerca de la inducción en la expresión de estas histonas en respuesta a situaciones de estrés o diferenciación. Los ratones mutantes nulos para H2AX presentan un retardo en el crecimiento. Al analizar fibroblastos embrionarios de estos ratones, se observó que crecen muy poco in vitro y que esto se debe a una disminución en el número de células en división; de hecho, el índice mitótico fue del 50% con respecto a las células de ratones salvajes (Celeste et al. 2002). En Tetrahymena thermophila, la fosforilacicón de H2AX es esencial para que la meiosis y la mitosis sean normales (Song et al. 2007). Nosotros observamos, en un 66 )#$! ensayo preliminar, que los taquizoitos que sobreexpresan H2AX (dos de tres clones) tendrían una mayor velocidad de replicación que los parásitos salvajes. Lo cual estaría de acuerdo con un rol de esta histona en la división celular. De confirmarse una correlación entre el incremento de H2AX y la tasa de replicación del parásito, se deberá ahondar en el mecanismo de acción que ésta presenta durante este proceso biológico. Además, basados en nuestros resultados, resta estudiar si esta sobreexpresión genera un rol protector frente a daño del ADN y si estos parásitos que sobreexpresan H2AX poseen mayor capacidad de diferenciación que los de la cepa salvaje. Es necesario un análisis exhaustivo de estos parásitos mutantes para entender mejor el rol funcional de esta variante en T. gondii. 67 Capítulo 4 ' )#$% /$0 4.1. Introducción El nucleosoma está compuesto por dos copias de cada una de los cuatro tipos de histonas: H2A, H2B, H3, and H4 (Luger et al. 1997). H2A y H2B forman dímeros, los cuales se asocian a ambos extremos de un tetrámero H3-H4. Las histonas H2A y H2B son notablemente diferentes en parásitos protozoarios que en mamíferos; de forma contraria a lo que ocurre con H3 y H4, las cuales mantienen sus similitudes a nivel de secuencia y modificaciones postraduccionales (Alsford and Horn 2004, Sullivan et al. 2006). En general, la región N-terminal de H2AZ en protozoos es más extensa comparada con la H2AZ de mamíferos. Además, como se mencionó en la discusión del capítulo 3, en P. falciparum ésta mostró estar acetilada en múltiples lisinas (Trelle et al. 2009) al igual que en T. thermophila (Ren and Gorovsky 2001). Estos dos parámetros claramente diferencian estas H2AZ de la de sus hospedadores humanos. La H2A canónica de T. brucei se encuentra peculiarmente hiperacetilada en su región C-terminal (Janzen et al. 2006) y posee una H2AZ que es esencial para la vida del parásito. TbH2AZ dimeriza con una H2B variante (TbH2Bv) y estos dímeros se encuentran ausentes de las regiones transcripcionalmente activas. Esta fue la primera documentación de una H2A capaz de interaccionar con una H2B no canónica (Lowell et al. 2005). La presencia de una H2B variante se ha descripto en varios protozoos (Lowell et al. 2005, Miao et al. 2006). Toxoplasma presenta un gen h2bv cuya expresión es igual en taquizoitos que en bradizoitos, mientras que la canónica presenta una expresión asociada al ciclo celular del parásito (Capítulo 2). De modo similar, P. falciparum, también presenta la variante H2Bv cuyos niveles de expresión permanecen invariables a lo largo del ciclo celular (Miao et al. 2006). Recientemente, se observó que PfH2Bv se encuentra acetilada, mientras la H2B canónica no, sugiriendo que estas dos histonas poseen roles diferentes (Trelle et al. 2009). Los dímeros H2A-H2B son intercambiados por dímeros H2AZ-H2B mediante la acción de un complejo remodelador de la cromatina ATP-dependiente de la familia SWI/SNF (Mizuguchi et al. 2004). La localización genómica de H2AZ en levaduras fue estudiada por varios grupos (Guillemette et al. 2005, Raisner et al. 2005, Zhang et al. 2005). En todos los casos, observaron que se encuentra preferencialmente en las regiones promotoras inactivas. Raisner y colaboradores (Raisner et al. 2005) examinaron la localización de H2AZ a una resolución de mononucleosoma y encontraron una fuerte preferencia de H2AZ por ser incorporada en los nucleosomas que flanquean la “región libre de nucleosomas” (Nucleosome free región, NFR). Sin embargo, no encontraron 69 )#$% /$0 correlación entre promotores activos e inactivos. La región NFR usualmente se encuentra 200 pb río arriba del primer codón e incluye el sitio de iniciación de la transcripción de casi todos los genes (Yuan et al. 2005). Sin embargo, Millar et al. (Millar et al. 2006) observó que si bien H2AZ se encuentra preferencialmente asociada a genes reprimidos, su forma acetilada está enriquecida en promotores de genes activos. Se conocen muy pocas modificaciones postraduccionales de H2AX, ya que su estudio se ha focalizado en la función de la fosforilación del motivo C-terminal característico. Sin embargo, recientemente se describió en células humanas que la radiación ionizante induce la acetilación de H2AX en K5 (mediada por TIP60), lo cual permite que la misma sea ubiquitinada y liberada de la cromatina dañada (Ikura et al. 2007). Por otro lado, se describió que el complejo FACT (formado por Spt16 y SSRP1) facilita la incorporación y la disociación de H2AX de los nucleosomas (Heo et al. 2008). La distribución genómica de H2AX no ha sido estudiada. La creencia general es que esta variante se encuentra distribuida de manera homogénea dentro del núcleo, formando foci discretos según se observa por microscopía 4Pi (Bewersdorf et al. 2006). Toxoplasma posee tres histonas H2A: H2A1, H2AX y H2AZ y dos histonas H2B: una canónica H2Ba y una variante H2Bv. Por lo tanto nos propusimos estudiar cómo interaccionan estas H2As y H2Bs para componer los nucleosomas, analizar el estado de acetilación de las mismas y/o su proximidad a histonas acetiladas; como también su localización en el genoma. 70 )#$% /$0 4.2. Resultados 4.2.1 Interacciones entre las histonas H2As y H2Bs Como se describió previamente, Toxoplasma posee una H2B variante, la cual es la mayoritaria. Como consecuencia, es posible que este parásito presente arreglos de nucleosomas novedosos. Con el fin de examinar las interacciones entre las histonas H2A y H2Bv, se realizaron coinmunoprecipitaciones (coIP) seguidas de Western blot con los anticuerpos específicos generados en el laboratorio. Figura 4.2.1. Inmunoprecipitaciones de las histonas H2A y H2Bv. Se realizaron coinmunoprecipitaciones con α-H2Bv (A), α-H2AX (B), α-H2A1 L1 (C) y α-H2AZ (D). El material inmunoprecipitado con los anticuerpos específicos (IP) y sus sueros preinmunes (Preinm), junto con el 1% del lisado total (Input, IN) y el 1% del lisado no unido al anticuerpo (NU), fueron analizados por Western blot con el anticuerpo utilizado para realizar la IP, respectivamente. Al igual que el mismo anticuerpo unido a la proteína A/G plus (PA+Ac). Las flechas indican las histonas H2Bv (A), H2AX (B), H2A1 (C) y H2AZ (D) de T. gondii. Primero se establecieron las condiciones óptimas de inmunoprecipitación para cada anticuerpo. Se analizó la presencia de la histona en el lisado antes (Input) y 71 )#$% /$0 después (no unido) de la incubación con el anticuerpo, así como también en la fracción inmunoprecipitada (IP) (Figura 4.2.1). Como control, se utilizaron los anticuerpos unidos a la proteína A y se realizaron inmunoprecipitaciones usando los sueros preinmunes. Una vez establecidas estas condiciones, se procedió al estudio de las interacciones entre histonas. Para analizar la interacción entre ellas dentro del nucleosoma, los taquizoitos fueron permeabilizados con digitonina y luego tratados con endonucleasa micrococcal (MNasa), con el fin de obtener una solución con > 95% de mononucleosomas (Figura 4.2.2A). A continuación se llevaron a cabo las IPs con αH2AX, α-H2AZ y α-H2Bv. Cada una se analizó por Western Blot con los otros dos anticuerpos y con anti-SAG1 como control de contaminación (Figura 4.2.2B). Se encontró que H2Bv es capaz de formar dímero con H2AZ, pero no con H2AX. Del mismo modo, H2AZ dimeriza con H2Bv mientras que H2AX no lo hace (Figura 4.2.2B). A su vez, H2AZ no coinmunoprecipita con H2AX, por lo que los taquizoitos no poseen nucleosomas donde uno de los dímeros H2A-H2B contenga H2AX y el otro H2AZ (Figura 4.2.2B). Figura 4.2.2. Interacción entre variantes de H2A y H2B. A. Los taquizoitos fueron lisados con MNasa (L1, L2 y L3). Un 10% de la muestra fue utilizado para extraer su ADN y analizarla por gel de agarosa 1,5% y visualizada con bromuro de etidio. A la derecha se observa un marcador de peso molecular (M). B. Se realizaron inmunoprecipitaciones (IP) con los anticuerpos detallados en la parte superior del panel. Luego fueron analizadas por Western blot (WB) con los anticuerpos detallados a la izquierda del panel. El 1% de lisado (IN) fue analizado en paralelo para determinar la presencia de la proteína en el lisado previo a la inmunoprecipitación. Los mismos experimentos de coIP también se llevaron a cabo utilizando taquizoitos sonicados. De este modo, se obtienen fragmentos de ADNg entre 100 y 600 pb (Figura 4.2.3A); los cuales contienen desde 1 a 4 nucleosomas. Como consecuencia, bajo estas condiciones se analiza lo que ocurre en estos pequeños fragmentos de cromatina. Se observó que H2AX no coinmunoprecipita con H2AZ ni con H2Bv. Del 72 )#$% /$0 mismo modo, H2AZ y H2Bv interaccionan entre si, pero ninguna coinmunoprecipita con H2AX (Figura 4.2.3B). Indicando que los nucleosomas conteniendo H2Bv-H2AZ no se encuentran adyacentes a los que contienen H2AX. Además se analizó la asociación de estas histonas con H2A1, utilizando el anticuerpo específico α-H2A1 L1. En este caso se observó que todas las variantes coinmunoprecipitaban con H2A1 (Figura 4.2.3B). Por lo que esta histona canónica se encuentra presente en las zonas donde están H2Bv-H2AZ como H2AX. Por lo tanto, podríamos pensar que H2A1 presenta una distribución más homogénea. * * * * * * Figura 4.2.3. Interacciones entre H2As y H2Bs. A. Análisis de los fragmentos de ADNg obtenidos luego de la sonicación de los parásitos en gel de agarosa 1%. L1, L2 y L3 son tres lisados utilizados para realizar inmunoprecipitaciones. M es el marcador de peso molecular 1 kb plus. B. Se realizaron inmunoprecipitaciones (IP) de lisados obtenidos por sonicado con los anticuerpos detallados a la izquierda del panel. Luego estos fueron analizados por Western blot (WB) con los anticuerpos detallados en la parte superior del panel; en cada caso se analizó el material inmunoprecipitado (IP) y el 1% del lisado (IN). Las flechas indican las bandas detectadas por cada anticuerpo. (*) indica las bandas correspondientes a la cadena liviana del anticuerpo utilizado para realizar la IP. 4.2.2 H2As y H2Bv: acetilación y asociación con histonas acetiladas Se ha descripto exhaustivamente la localización de histonas acetiladas en la cromatina activa (Berger 2002). Con el objetivo de determinar si las histonas H2A y H2Bv se encuentran asociadas a comatina activa, se realizaron dos estrategias diferentes basadas en inmunoprecipitaciones con parásitos sonicados. Por un lado se inmunoprecipitaron las histonas, H2AX, H2AZ y H2Bv con sus respectivos anticuerpos policlonales y los inmunocomplejos se analizaron por Western blot con anti-H3 acetilada (α-AcH3), la cual se sabe que se encuentra asociada a cromatina activa en T. gondii 73 )#$% (Saksouk et al. 2005, Gissot et al. /$0 2007). Las tres histonas variantes coinmunoprecipitaron con AcH3 (Figura 4.2.4A). Por otro lado, se realizó una inmunoprecipitación con anti-lisina acetilada (α-LysAc) y se evaluó la presencia de las H2As y H2Bv en los inmunocomplejos por Western blot, utilizando α-AcH3 como control positivo. H2AZ, H2A1 y H2Bv se detectan con intensidad similar a la observada para AcH3; mientras que H2AX requiere un tiempo prolongado de exposición para ser apenas detectada (Figura 4.2.4B). Si se comparan las intensidades de las bandas, se puede observar que el porcentaje de H2AZ, H2A1 y H2Bv inmunoprecipitado con respecto al 1% del lisado (IN) es menor, pero similar a lo observado para AcH3; mientras que el porcentaje de H2AX es más de diez veces menor (datos no mostrados). Como estas inmunoprecipitaciones fueron realizadas con cromatina sonicada y más de un nucleosoma es inmunoprecipitado, podemos concluir que si bien H2AX es capaz de inmunoprecipitar con AcH3, cuando se analiza el conjunto de histonas acetiladas y/o cercanas a histonas acetiladas es la minoritaria. Por lo tanto, podríamos decir que sólo H2AZ y H2Bv muestran una clara asociación con histonas acetiladas y/o se encuentran acetiladas. Figura 4.2.4. Relación entre las histonas H2As y H2Bv y la acetilación. Se utilizaron lisados de taquizoitos generados por sonicación para realizar coinmunoprecipitaciones (IP). A. Se realizaron IPs con los anticuerpos detallados en la parte superior de la figura. Se analizaron por Western blot (WB) con los mismos anticuerpos como control de que la IP funcionó correctamente (CTR) y con anti-H3 acetilada (α-AcH3). B. Se realizó una IP con anti-lisina acetilada (α-AcLys) y se la analizó por WB con los anticuerpos detallados a la izquierda del panel. En cada caso se analizó en paralelo el 1% del lisado antes de ser inmunoprecipitado (IN). 74 )#$% /$0 4.2.2 Asociación diferencial de H2A y H2B variantes a genes activos e inactivos Nuestros datos indican que H2AX y H2AZ/H2Bv se encuentran en nucleosomas diferentes y que podrían poseer diferentes niveles de acetilación y/o estar próximas a histonas acetiladas; ambos marcas de cromatina transcripcionalmente activa, entre otros roles (Kouzarides 2007). Para analizar lo que ocurre con mayor detalle, se realizaron experimentos de inmunoprecipitación de cromatina (ChIP) como se describe en el capítulo 6, empleando los anticuerpos α-H2AX, α-H2AZ y α-H2Bv seguidos de PCR en tiempo real, en un análisis cuantitativo (qPCR). En paralelo se realizaron ChIP-qPCR con α-AcH3 y α-H4K20me1 (histona H4 monometilada en K20) como controles de eucromatina y heterocromatina, respectivamente (Saksouk et al. 2005, Sautel et al. 2007). Se utilizaron oligonucleótidos que amplifican en la región promotora de un gen activo constitutivamente (β-tubulin), de un gen de expresión específica en taquizoito (sag1) y de dos genes específicos de bradizoitos, los cuales se encuentran reprimidos en el estadio de taquizoito (ldh2 y bag1). Estos oligonucleótidos fueron diseñados para amplificar secuencias localizadas 500 - 700 pb río arriba del codón de inicio (ver tabla 3 en capítulo 6). Gissot y colaboradores encontraron que a esa distancia del gen se observa un enriquecimiento de las histonas modificadas postraduccionalmente (Gissot et al. 2007). Si bien H2AX, H2AZ y H2Bv coinmuoprecipitan con todas las regiones genómicas analizadas, estos estudios indican que H2AX y H4K20me1 se encuentran enriquecidas en los genes reprimidos ldh2 y bag1 en comparación con los genes activos β-tubulin y sag1. De forma inversa, H2AZ, H2Bv y AcH3 se encuentran enriquecidas en genes activos en relación con los inactivos (Figura 4.2.5A). Estos datos sugieren que H2AZ y H2AX podrían estar regulando el estado de la cromatina de forma opuesta. 4.2.4 H2AX se encuentra asociada a TgIRE, un elemento repetitivo al extremo de los cromosomas La asociación de H2AX con promotores inactivos promueve la idea de que esta histona podría contribuir con regiones genómicas transcripcionalmente reprimidas como ADN silente. Con el objetivo de identificar regiones libres de genes en Toxoplasma, se utilizaron elementos repetitivos previamente descriptos para realizar una búsqueda en el genoma del parásito (ToxoDB). Cuando se realizó la búsqueda con los elementos 75 )#$% /$0 repetitivos sat350 y sat680 (Clemente et al. 2004) se encontraron secuencias poco conservadas o incompletas (datos no mostrados). En cambio, al utilizar TgIRE (Echeverria et al. 2000) se encontraron secuencias con alta homología presentes en 9 de los 14 cromosomas (Figura 4.2.5B). El tamaño de este elemento repetitivo varía entre cromosomas. En la mayoría de los casos es de aproximadamente 1900 pb, excepto en los cromosomas IV y V, en los cuales presenta una deleción entre las bases 240/270 y 1402 (datos no mostrados). La búsqueda de productos de expresión dentro de TgIRE en ToxoDB devolvió cuatro ESTs, que se encuentran sólo en 3 cromosomas. Tres de ellos (TIGR EST Assemblies: N61085, TC35464 and CK736714) se encuentran en los cromosomas Ia y XI, mientras que el restante (TIGR EST Assemblies: TC39371) en el cromosoma X. Sin embargo, ninguno de ellos corresponde a un gen o a una proteína putativa, ni existen evidencias de péptidos por espectrometría de masa. Teniendo en cuenta estos resultados in sílico, propusimos a TgIRE como una región silenciada del ADN. De acuerdo con esta idea, H4K20me1 se encuentra enriquecida en esta región, mientras que AcH3 está completamente ausente cuando se las analiza por ChIP-qPCR. En correlación con lo observado previamente, H2AX se encuentra enriquecida en TgIRE mientras que H2AZ y H2Bv no lo están (Figura 4.2.5A). Estos resultados son consistentes con la idea de que H2AX es una histona variante que se encuentra asociada con genes o regiones del genoma silenciadas en T. gondii. 76 )#$% /$0 Figura 4.2.5. Distribución genómica de las variantes H2A y H2B. A. Se realizaron ensayos de Inmunoprecipitación de la cromatina (ChIP) con los anticuerpos α-H2AX, α-H2AZ y α-H2Bv (gráficos a la izquierda de la figura). Además, se utilizaron los anticuerpos comerciales α-H4K20me1 y α-AcH3 (gráficos a la derecha de la figura) como controles de heterocromatina y eucromatina, respectivamente. El ADN inmunoprecipitado fue analizado por qPCR amplificando secuencias río arriba de los promotores de sag1, β-tubulina (tub), bag1 y ldh2 y dos zonas de TgIRE localizadas a los 100 pb (IRE100) y 600 pb (IRE 600). B. Representación gráfica de la localización de TgIRE (casillas blancas) en los cromosomas de T. gondii. Los números romanos indentifican el cromosoma, mientras que los número cardinales indican la posición de TgIRE en cada cromosoma. 77 )#$% /$0 4.3 Discusión Al analizar la composición de los nucleosomas de T. gondii, se realizó un hallazgo interesante: la histona H2AX no es capaz de formar dímeros con H2Bv mientras que la H2AZ si lo hace. Además, estas dos H2A variantes no se encuentran en el mismo nucleosoma; indicando que las histonas H2AX y H2AZ/H2Bv tendrían diferentes roles en la dinámica de la cromatina. Se podría pensar que es lo mismo que ocurre en T. brucei, donde la H2Bv interacciona con H2AZ pero no con H2A (Lowell et al. 2005), ya que este parásito no posee H2AX. Como los organismos eucariotas superiores no poseen H2B variantes, es probable que los parásitos protozoarios presenten un mecanismo de regulación de la cromatina particular basado en la incorporación de H2AZ y H2Bv en los nucleosomas. De acuerdo con la idea de que las variantes de histonas H2A y H2B tendrían características particulares y diferencias en la modulación de la cromatina, se observó por ChIP-qPCR que H2AZ, H2AX y H2Bv se encuentran en las regiones río arriba de genes activos e inactivos; sin embargo, el enriquecimiento de cada una de ellas es diferente. La H2AX se encuentra enriquecida en genes inactivos como también en regiones genómicas silentes (TgIRE) mientras que la H2AZ y la H2Bv se encuentran enriquecidos en los promotores de genes activos. Los estudios de localización genómica de H2AZ realizados hasta el momento en levadura, revelan que ésta se encuentra asociada preferentemente a promotores inactivos (Guillemette et al. 2005, Li et al. 2005b, Lowell et al. 2005, Pattenden et al. 2005, Zhang et al. 2005), a excepción de Raisner et al (Raisner et al. 2005), que no encontró correlación entre la expresión de los genes y la presencia de H2AZ en sus promotores. Sin embargo, recientemente se publicaron tres estudios, uno en linfocitos T de humanos (Barski et al. 2007), otro en C. elegans (Whittle et al. 2008) y el tercero en T. brucei (Siegel et al. 2009), en los que encontraron una correlación positiva entre la presencia de H2AZ en los promotores y la transcripción de esos genes. En particular, en T. brucei se observó que H2AZ y H2Bv se encuentran en los mismos nucleosomas, los cuales fueron detectados en los probables sitios de inicio de la transcripción por la ARN polimerasa II (transcription start sites, TSS). Además, observaron que estos nucleosomas son menos estables que los que contienen las correspondientes histonas canónicas, sugiriendo que tanto H2AZ como H2Bv contribuyen a una estructura de la cromatina más abierta en los TSS. Estos resultados se correlacionan con los encontrados en Toxoplasma, donde H2AZ y H2Bv se encuentran en el mismo nucleosoma y se localizan en regiones activas. Una vez más, en 78 )#$% /$0 Tripanosomátidos y Apicomplexas, las variantes H2AZ y H2Bv estarían modulando el estado de la cromatina a través de un mecanismo novedoso. En este trabajo se observó también que H2AZ y H2Bv coinmunoprecipitan junto con AcH3. Esto significa que estas variantes se encuentran interaccionando con AcH3 o en nucleosomas cercanos. Asimismo, estas histonas inmunoprecipitaron con α-AcLys, indicando que estas misma podrían estar acetiladas, como se vio en H2AZ y H2Bv de P. falciparum (Trelle et al. 2009) y/o que están estrechamente relacionadas a histonas acetiladas. Todos estos datos concuerdan a su vez con el hecho de que se encuentran enriquecidas en promotores activos. Por el contrario, H2AX es capaz de coinmunoprecipitar con AcH3, pero el hecho de que durante la inmunoprecipitación con α-AcLys, la presencia de esta histona fue casi indetectable, indicaría que no está acetilada ni se encuentra estrechamente asociada con otras histonas acetiladas. Esto concuerda con el hecho de que H2AX está ligada a regiones silentes del ADN. La histona H3 acetilada que coinmunoprecipita con H2AX puede representar a la porción de AcH3 que se encuentra en los bordes del ADN silenciado o que colocaliza con la pequeña fracción de H2AX que es detectada en las regiones activas por ChIP. Sin bien nuestros estudios muestran que H2AZ y H2Bv están fuertemente asociadas a regiones activas de la cromatina y que H2AX está asociada a regiones silentes, también muestran que las tres variantes de Toxoplasma se encuentran localizadas en genes activos e inactivos. Una posible explicación sería que las modificaciones postraduccionales sean los responsables de estos enriquecimientos. Por ejemplo, que H2AZ se encuentre acetilada en los promotores activos, como observó Millar et al en levaduras (Millar et al. 2006), siendo esta forma la más abundante; mientras que la H2AZ que se encuentra en regiones silenciadas esté monoubiquitinada como en mamíferos (Sarcinella et al. 2007). Sería interesante realizar diversos experimentos para determinar las modificaciones postraduccionales de las histonas H2A y H2B en T. gondii y conocer su contribución al estado de la cromatina. Un hallazgo de este trabajo que consideramos muy importante es la asignación de un nuevo rol a H2AX, no relacionado con el daño del ADN, sino como posible modulador de la cromatina activa. Incluso, se puede ligar este hallazgo al hecho de que durante la diferenciación a bradizoito se observa un aumento en la expresión de esta histona variante (Capítulo 3), con su asociación a regiones silenciadas, basados en que los bradizoitos presentan un mayor número de genes reprimidos (Roos D. comunicación personal). Esto estaría sugiriendo que H2AX, al menos en este parásito, posee una función asociada al silenciamiento de la cromatina mucho más extensa que lo observado 79 )#$% /$0 en mamíferos; donde γH2AX es necesaria para la inactivación de los cromosomas X e Y durante la meiosis en machos (Fernandez-Capetillo et al. 2003). Recientemente, Shechter et al (Shechter et al. 2009) encontró que Xenopus laevis tiene dos tipos de H2AX, una con el motivo C-terminal SQEY (igual que mamíferos), la cual es la mayoritaria en adultos y otra SQEF (igual que Toxoplasma) que constituye el 50% del total de H2A en oocitos y embriones tempranos pero es la minoritaria en estadios maduros. Los autores proponen que esta isoforma estaría involucrada en modular la respuesta celular frente a la rapidez de los ciclos celulares que ocurren de forma temprana durante el desarrollo. Es interesante destacar que esta H2AX-SQEF se encuentra fosforilada en células en activa replicación y que esta fosforilación más que estar asociada al ciclo celular estaría ligada a rearreglos en la cromatina. Por otro lado, en mamíferos se observó que la tirosina (Y) de H2AX-SQEY es capaz de ser fosforilada y estaría regulando la fosforilación de la serina (S) durante la reparación del daño del ADN (Xiao A 2009). Como la fenilalanina (F) no es un aa. fosforilable, este mecanismo no puede tener lugar para el caso de H2AX-SQEF. Nos preguntamos si H2AX-SQEY es más importante en el buen control de la reparación del ADN, mientras que H2AX-SQEF lo es durante el desarrollo. Si fuera así, hasta podría plantearse que estos dos motivos funcionan como plataformas alternativas para el ensamblado de una maquinaria modeladora de cromatina específica para cada contexto; y que en los distintos organismos H2AX ha evolucionado en base a las necesidades de cada uno. Dado que todos estos hallazgos son muy recientes, suponemos que en los próximos años se irán dilucidando estos cuestionamientos, donde esperamos que T. gondii, como modelo protozoario, pueda aportar datos interesantes al respecto. En el afán de encontrar regiones silenciadas en T. gondii, analizamos en detalle a TgIRE, secuencia repetitiva previamente descripta en el laboratorio (Echeverria et al. 2005). Un análisis más exhaustivo mostró que se encuentra formando parte de una región que comprende entre 10 a 30 kpb, la cual presenta sintenia entre los 9 cromosomas que presentan este elemento. A esta región la denominamos región conservada en el extremo cromosómico (ChCER, por “chromosome conserved end region”). Tanto en P. falciparum como en Trypanosoma ssp. se han descripto regiones subteloméricas, donde se encuentran los genes var y VSG respectivamente, los cuales son responsables de la variación antigénica de estos parásitos (Van der Ploeg 1991, Figueiredo et al. 2000). En P. falciparum, se encuentran compuestas por elementos repetitivos y son las responsables de que los telómeros formen clusters en la región periférica del núcleo, lo cual favorece los rearreglos entre los genes var (Figueiredo et al. 2002). Por otro lado, en T. brucei se observó que éstas secuencias están enriquecidas en 80 )#$% /$0 una variante específica de la histona H3, H3V (Lowell and Cross 2004). Toxoplasma no posee genes homólogos a los genes var o VSG; por lo que esta región conservada sólo en uno de los extremos de ciertos cromosomas es sumamente llamativa y poco común. Nosotros proponemos que ChCER podría tener arreglos de nucleosomas particulares y una función importante en el anclaje de estos cromosomas durante algún mecanismo de regulación celular, como podría ser la replicación del parásito. Actualmente se están encarando estudios para dar luz a estas preguntas. 81 Capítulo 5 ) )#$1 ) El conocimiento acerca de las histonas es esencial para comprender cómo la transcripción, la replicación y otros procesos celulares operan en T. gondii. Nosotros estudiamos las histonas H2A y H2B, que no habían sido estudiadas hasta el momento en este parásito. 5.1. T. gondii tiene una H2B canónica y una H2B variante Observamos que este parásito contiene tres H2Bs en su genoma: H2Ba, H2Bb y H2Bv, sin embargo H2Bb no se expresa en taquizoito ni bradizoito. Los niveles de transcripto de H2Ba son menores que los de H2Bv en taquizoitos y estos niveles disminuyen aún más en el estadio de bradizoito (arrestado en G0); indicando por esto último que es una histona canónica. En cambio, los niveles de transcripto como los de proteína de H2Bv permanecen constantes en los dos estadios, confirmando su calidad de variante. En correlación con la hipótesis de que H2Bv es la H2B mayoritaria, fue la única H2B detectada por espectrometría de masa de histonas purificadas. El análisis filogenético (Neighbor joining) de las histonas H2B muestra que se agrupan en dos linajes diferentes: uno conteniendo las H2B canónicas y el otro la H2B variante (H2Bv). Estos dos clados se encuentran en todos los parásitos del phylum Apicomplexa analizados (también ver (Sullivan et al. 2006)), sugiriendo que la duplicación de este gen ocurrió previamente a la divergencia del phylum . Además, las H2B variantes parecen ser más conservadas que las H2B canónicas, indicando diferentes tasas de evolución de estos genes. 5.2. T. gondii tiene una H2A canónica y dos H2As variantes: H2AX y H2AZ Por otro lado, observamos que T. gondii presenta tres H2A en su genoma: H2A1, H2AZ y H2AX; las cuales se transcriben y traducen en taquizoitos. H2A1 es una histona canónica con una expresión asociada a la fase S del ciclo celular, ya que sus niveles en bradizoitos maduros son indetectables. H2AZ es la histona H2A minoritaria y su expresión a nivel de mRNA no se ve afectada por la diferenciación a bradizoito, ni por la presencia de estrés oxidativo o alquilante. Los niveles de ARNm de h2aX son mayores en bradizoitos que en taquizoitos, confirmando su calidad de variante. H2AX se encuentra fosforilada en el motivo característico C-terminal y la fosforilación se incrementa en presencia de H2O2. Esto indica que la fosforilación de H2AX frente a daño del ADN se encuentra conservada en este parásito. Además, su sobreexpresión provoca una mayor 83 )#$1 ) velocidad de replicación, lo cual estaría dando un indicio a cerca de su función en el parásito; aunque hacen falta mayor número de estudios para esclarecer el rol que desempeña. 5.3. La expresión de H2AX se incrementa ante estrés oxidativo y durante la diferenciación Hasta la fecha no hay datos que muestren que existiría una regulación de la expresión de h2aX. En T. gondii, esta histona aumenta su expresión en bradizoitos y frente a estrés oxidativo, pero permanece invariable durante estrés térmico; indicando que no se sobreexpresa ante cualquier tipo de estrés. 5.4. Las variantes H2Bv, H2AZ y H2AX componen distintos nucleosomas y participan diferencialmente en la modulación de la cromatina activa La presencia de variantes H2A y H2B le confiere al parásito la posibilidad de presentar nucleosomas especializados: H2AZ dimeriza con H2Bv, pero esta última no lo hace con H2AX, al mismo tiempo que H2AX no está presente en el mismo nucleosoma que H2AZ. La formación de dímeros H2A-H2B para formar los nucleosomas no es aleatoria. Toxoplasma estaría regulando ciertos procesos celulares a través de un mecanismo epigenético basado en la dimerización de H2Bv con H2AZ y no con H2AX, donde H2Bv y H2AZ se encuentran enriquecidos en los promotores de genes activos. Por otro lado, H2AX está predominantemente en cromatina silenciada y sus niveles de transcripto aumentan considerablemente en bradizoitos, los cuales se espera que tengan mayor número de genes silenciados. Se podría pensar en un mecanismo epigenético diferente, basado en que H2AX estaría involucrada en el silenciamiento de genes y por lo tanto en la diferenciación del parásito. Sería muy interesante dilucidar si el aumento de H2AX estimula el silenciamiento de genes y la diferenciación a bradizoito o si la diferenciación a bradizoito estimula la sobreexpresión de H2AX para poder silenciar varios genes. 5.5. Modelo propuesto Basados en los resultados obtenidos proponemos el siguiente modelo: los dímeros H2AZ-H2Bv se encuentran regulando de algún modo la transcripción de genes; 84 )#$1 ) están asociadas a AcH3 y es probable que ellas también se encuentren acetiladas. Por otro lado, H2AX es importante para el silenciamiento de genes; encontrándose enriquecida en las mismas regiones que H4K20me1. Todas las funciones que se le han adjudicado a H2AX se encuentran relacionadas con su forma fosforilada, podría ser que en este caso también se encuentre fosforilada. En todos los casos vimos que si bien hay un claro enriquecimiento de H2AZ-H2Bv en promotores activos y de H2AX en promotores inactivos o zonas silenciadas, las tres se encuentran presentes en todas las regiones. Esto podría estar dado por modificaciones postraduccionales particulares para cada caso, aún no descriptas. Figura 5.1. Modelo propuesto. Composición nucleosomal de los promotores en T. gondii. 85 Capítulo 6 * )#$& * 6.1. Cultivo de parásitos T. gondii comprende una población clonal compuesta por tres genotipos diferentes denominados tipo I, tipo II y tipo III. Estas poseen un alto grado de similitudes morfológicas, antigénicas y moleculares. Sin embargo, las cepas tipo I (como RH y CT1) son altamente virulentas, que en baja cantidad inducen la muerte de ratones SwissWebster durante la fase aguda de la infección. Mientras que las cepas tipo II (como ME49 y PRU) y III (como COUG y VEG) poseen una virulencia relativa y permiten que se establezca una infección crónica. Los experimentos fueron realizados utilizando tres cepas diferentes de T. gondii: RHΔuprt, RHΔhxgprt (tipo I) y PK (tipo II). Para la obtención de taquizoitos todas las cepas fueron cultivados in vitro en condiciones estándar: se infectaron células HFF (“Human Foreskin Fibroblast”- fibroblastos humanos de prepucio) en monocapa y se incubaron con medio DMEM (Gibco) suplementado con 10% de suero fetal bovino (SFB) a 37°C y 5% de CO2. Los parásitos RHΔhxgprt son deficientes en la enzima hipoxantina xantina guanina fosforibosil transferasa. Esta enzima permite seleccionar positiva y/o negativamente los parásitos (ver sección “Sobreexpresión de proteínas en Toxoplasma” y (Donald et al. 1996)). Para la obtención de bradizoitos se utilizaron dos sistemas diferentes, uno in vitro y el otro in vivo. In vitro: Se utilizaron parásitos RH knock-out para la enzima uracil fosfo-ribosil transferasa (RHΔuprt), la cual normalmente cataliza la formación de UMP (uridina monofosfato) a partir de uracilo. Esta cepa mutante se obtuvo a partir de parásitos RHΔhxgprt. Esta enzima no es esencial ya que Toxoplasma contiene la vía de síntesis de novo de pirimidinas. Sin embargo, como el CO2 es necesario para que esta vía pueda ocurrir, cuando se los cultiva en ausencia de CO2 presentan un crecimiento más lento y una frecuencia de diferenciación 3-4 veces mayor (Bohne and Roos 1997). Se infectaron células HFF en monocapa (1 parásito/célula), se incubaron 10 min en hielo y luego 1 h en condiciones estándar de cultivo de taquizoitos para facilitar la invasión. A continuación se incubaron en condiciones de bradizoito: en medio MEM o RPMI (Gibco) suplementado con 5% de SFB y 25mM HEPES a pH 8,1 en concentraciones de CO2 ambiente (0,03%). Al cabo de 4 días, se levantó la monocapa y se pasó las suspensión por una aguja 27G con el fin de romper la pared de los quistes y liberar los bradizoitos. El porcentaje de bradizoitos fue analizado por inmunofluorescencia indirecta (IFA) utilizando la lectina de 87 )#$& * Dolichos biflorus (Boothroyd et al., 1997) que tiñe la pared del quiste y con α-BAG1 (bradizoyte antigen 1) que se encuentra en citoplasma de bradizoitos. In vivo: Se utilizaron parásitos PK, los cuáles son un clon derivado de la cepa Me49 (tipo II). Se extrajeron cerebros de ratones C3H un mes post-infección via oral y se homogeneizaron en 1 ml de tampón fosfato (PBS). Mediante una centrifugación a 1800 rpm con Dextran 30% (Sigma) en PBS, se obtuvo una fracción enriquecida en quistes. Estos fueron visualizados y contados al microscopio óptico. Los taquizoitos y bradizoitos obtenidos de cultivos in vitro fueron purificados de los restos celulares al ser pasados por un filtro de 3 mm de poro (Nucleopore). 6.2. Análisis de secuencia Las búsquedas en base de datos, el análisis de comparación de secuencias y el análisis de los dominios putativos se realizaron utilizando los programas Blastn, Blastx, Blast 2 y Blast domain (www.ncbi.nlm.nih.gov/Blast). Las histonas H2A y H2B de Toxoplasma se identificaron realizando Blast con las secuencias de histonas descriptas para otros organismos en la base de datos de T. gondii: http://ToxoDB.org. La localización de las mismas en los cromosomas se analizó con Genome Browser de la misma base de datos. Los alineamientos y análisis de secuencia se realizaron con el programa Bioedit. Las estructuras secundarias se determinaron utilizando herramientas que se encuentran en http://toolkit.tuebingen.mpg.de/sections/secstruct. 6.3. Análisis de Neighbor joining Se realizó un análisis de Neighbor joining (NJ) de la secuencia aminoacídica deducida de histonas H2A y H2B de T. gondii y otras histonas previamente descriptas utilizando el programa MEGA4 (Molecular Evolutionary Genetics Analysis Software version 4) como describe Tamura et al (Tamura et al. 2007). El soporte interno de las ramas se obtuvo mediante la opción bootstrap (búsqueda heurística) usando 1000 repeticiones. Las secuencias utilizadas para realizar el árbol de NJ de histonas H2B fueron: Pf: Plasmodium falciparum (AN: BU497652 y U14734), Pv: Plasmodium vivax (AN: CAA76839) Et: Eimeria tenella (AN: CD345163 y CD569058), Nc: Neospora caninun (AN: CF598462 y CF937965), Sn: Sarcocystis neuron (AN: CV193312 y CV194457), Ch: Cryptosporidium hominis (AN: XP_666723 y XP_666188). hTSH2B: human testis/sperm specific H2B (AN: AP397301). 88 )#$& * Por otro lado, las secuencias utilizadas para realizar el árbol de NJ de histonas H2A fueron: At, Arabidopsis thaliana (AN: AAL85051 y BAF00012), Dm, Drosophila melanogaster (AN: AAN11125 y AAF56631), Gl, Giardia lamblia (AN: AF139873); Hu, human (AN: AAN59958, P16104 y CAG33696), Pf, Plasmodium falciparum (AN: P40282 y CAB39069); Pyy, Plasmodium yoelii yoelii (AN: CAQ40903 y EAA15833); Sc, Saccharomyces cerevisiae (AN: P04909, P04910 y Q12692), Tg, Toxoplasma gondii, Tt, Tetrahymena thermophilus (AN: AAC37292, AAC37291 y CAA33554), Xl, Xenopus laevis (AN: AAA49769, AAH74188 y AAH74203). 6.4. Amplificación y clonado de las diferentes histonas Con el objetivo de amplificar y clonar los genes de las diferentes histonas H2A y H2B de T. gondii se realizaron ensayos PCR, utilizando como templado ADNg y ADNc. Para ello se colectaron taquizoitos RH frescos que rápidamente se solubilizaron en TRIzol (Invitrogen Life Technologies). Siguiendo las instrucciones del fabricante se extrajo ADNg y ARN. El ADNc se obtuvo mediante transcripción en forma reversa de 2 μg del ARN utilizando la enzima MMLV (InvitrogenTM, Life Technologies) y oligo(dT)12-18. La secuencia codificante (CDS) de cada una de las histonas fue amplificada utilizando oligonucleótidos específicos: H2Ba (H2Ba F- H2Ba R), H2Bb (H2Bb F- H2Bb R), H2A1 (H2A1 F- H2A1 R), H2AX (H2AX F- H2AX R) y H2AZ (H2AZ F- H2AZ R) (Tabla 1). Cada una de las reacciones se llevó a cabo en un volumen final de 20 μl, conteniendo 0,05 U/μl de Taq DNA polymerasa (Promega), 1,6 mM MgCl2, 200 μM de dNTP, 100 pM de cada oligonucleótido específico y 1 ȝl de templado. Las condiciones de las PCRs fueron las siguientes: 1 ciclo de desnaturalización: 5 min a 95ºC, 35 ciclos de amplificación que incluían: desnaturalización a 94ºC por 35 seg, apareamiento a la temperatura óptima determinada para cada par de oligonucleótidos (Tabla 1) por 45 seg y polimerización a 72ºC por 45 seg; seguidos de un último paso de elongación a 72ºC por 10 min. Las amplificaciones resultantes se visualizaron por electroforesis en gel de agarosa 1% con bromuro de etidio. Los amplicones de tamaño esperado fueron extraídos del gel con el kit Qiaex II (Qiagen). Cada uno de los productos de PCR purificado fue clonado en el vector bacteriano pGEM®-T Easy (Promega) siguiendo los protocolos suministrados. Finalmente, se transformaron bacterias E. coli (XL1-Blue) termocompetentes y se crecieron en placas de LB-Agar conteniendo Ampicilina y X-Gal/IPTG. Las colonias blancas fueron picadas y crecidas en medio líquido LB/ampicilina. El ADN plasmídico fue extraído empleando el método de lisis alcalina y posteriormente secuenciado. Las secuencias obtenidas fueron publicadas en GenBank: H2Ba (AN: 89 )#$& * DQ118972), H2Bv (AN: AF406555), H2A1 (AY573602), H2AX (AY631392) y H2AZ (AF502246). Table1. Oligonucleótidos utilizados en clonado y expresión de las histonas. Ta (°C) Descripción Producto (pb desde el ATG) Nombre Secuencia H2Ba F H2Ba R 5’-GGATCCATGGTGGCCAAGAAGTCC 5’-GGTACCGAAGTGTAAACTGCCGAGAC 68 CDS de h2ba 1 a 446 H2Bb F H2Bb R 5’-GGATCCATGGTGGCCAAGAAGTCC 5’-GGTACCTCCATTGCAAAGCTGCCGAG 56 CDS de h2bb 1 a 407 H2Bv F H2Bv R 5’-GGATCCATGTCAGGGAAAGGTCCG 5’-GGTACCCTATGCACCAGAAGTCGT 64 CDS de h2bv 1 a 372 H2A1 F H2A1 R 5’-GGATCCATGTCGGCCAAAGGCAAGG 5’-GGTACCTTACTGAGACTTCTTGCCCTTG 58 CDS de h2a1 1 a 390 H2AX F H2AX R 5’-GGATCCATGTCCGCCAAAGGTGCAGG 5’-GGTACCCACCAGACAGAATGGCTATCCT 58 CDS de h2aX 1 a 663 H2AZ F H2AZ R 5’-GGATCCATGGACGGAGCTGCAAAGT 5’-AAGCTTGAGCGACTTCTCGTGGAAAG 64 CDS de h2aZ 1 a 511 αTUB F αTUB R 5’-GAGCTCTTCTGCCTGGAA 5’-ATTTGGAGCCCACCGTCG 60 CDS de α-tubulina 64 a 223 qRT-H2A1 F qRT-H2A1 R 5’-AGGACGATGCAGCACACAAC 5’-CCATTAAAAGCCGCTTACGG 3’UTR de h2a1 622 a 715 qRT-H2AX F qRT-H2AX R 5’-CCCGCCTTTTAGGCTTCACT 5’-TGGTGAATTCGGCTCTTTGG 3’UTR de h2aX 615 a 708 qRT-H2AZ F qRT-H2AZ R 5’-TCTGGAGGGACAAACATGGG 5’-AAAAAACACGCACCTGACCC 3’UTR de h2aZ 581 a 674 qRT-TUB F qRT-TUB R 5’-ATGTTCCGTGGTCGCATGT 5’-TGGGAATCCACTGAACGAAGT CDS de β-tubulina 945 a 1039 CDS de bag1 536 a 630 5’-ACAATGGCCCAGGCATTCT 5’-CAATAAACATATCGTGAAGCCCATA CDS de ldh2 750 a 844 5’-TTAAGTGAGAACCCGTGGCAG 5’-GCTTTTTGACTCGGCTGGAA CDS de sag1 696 a 789 qRT-BAG1 F 5’-TGAGCGAGTGTCCGGTTATTT qRT-BAG1 R 5’-TAGAACGCCGTTGTCCATTG qRT-LDH2 F qRT-LDH2 R qRT-SAG1 F qRT-SAG1 R Ta: temperatura de apareamiento. qRT: RT-PCR en tiempo real. F: sentido. R: antisentido. GGATCC: secuencia reconocida por BamHI. GGTACC: secuencia reconocida por KpnI. AAGCTT: secuencia reconocida por HindIII. 6.5. Análisis de expresión H2Bs – La expresión de las 3 histonas H2B en taquizoitos y bradizoitos fue analizada por ensayos de RT-PCR semicuantitativa. Las reacciones de amplificación por 90 )#$& * PCR se llevaron a cabo utilizando ADNg y ADNc de taquizoitos y bradizoitos in vivo como templado en las mismas condiciones de reacción anteriormente descriptas. Los oligonucleótidos utilizados fueron: H2Ba F y R, H2Bb F y R y H2Bv F y R (Tabla 1). Se preparó un tubo de reacción con 120 μl para cada una de las histonas H2B y se los alicuotó en 6 tubos que fueron tomados a los 22, 25, 27, 28, 30 y 33 ciclos. Por un lado, se analizó la expresión relativa de cada uno de estos genes en taquizoitos comparando las curvas de amplificación de ADNc con respecto a ADNg, teniendo en consideración que existe una única copia de cada uno de estos genes en el genoma. Por otro lado, se compararon los niveles de ARN mensajero de cada histona en taquizoitos con respecto a bradizoitos. En este caso se tomaron las muestras a los números de ciclos correspondientes a la fase lineal de la curva de amplificación en taquizoitos. Para asegurar que iguales cantidades de ADNc de cada estadio fue comparado, se utilizaron oligonucleótidos que amplifican α-tubulina de T. gondii (Tabla 1) como control. Todos los productos de PCR fueron resueltos en un gel de agarosa 1,5 % y teñido sumergiendo el mismo en una solución de bromuro de etidio 1 %, para eliminar variaciones en el teñido de los geles. Finalmente, las imágenes fueron capturadas y digitalizadas para analizar la intensidad de las bandas con el programa “Gel-Pro analyzer” (Media Cybernetics). Estos experimentos fueron realizados por triplicado. El análisis estadístico fue llevado a cabo con el programa Prism4 version 3.0 (GraphPad Software, San Diego, CA, USA). Como control negativo se llevaron a cabo PCRs con cada par de oligonucleótidos utilizando como templado ADNc de células HFF sin infectar y de cerebro de ratón. H2As – La expresión de las histonas H2A se analizó y comparó en taquizoitos y bradizoitos in vivo e in vitro. En el primer caso (in vivo), se realizó mediante ensayos de RT-PCR semicuantitativa como se describió para H2B, utilizando los oligonucleótidos H2A1 F y R, H2AX F y R y H2AZ F y R (Tabla 1). Mientras que para taquizoitos y bradizoitos in vitro, se realizó por PCR en tiempo real (qRT-PCR). En este último caso, el ARN total de cada uno de los estadios fue extraído utilizando RNAeasy Mini Kit (Qiagen). El ADNc se obtuvo por transcripción en forma reversa de 2 μg de ARN utilizando Omniscript RT Kit (Qiagen), siguiendo las indicaciones del fabricante. Las reacciones de amplificación se llevaron a cabo en un volumen final de 25 μl conteniendo SYBR Green PCR Master Mix (Applied Biosystems, CA), 0,5 μM de cada oligonucleótido específico y 1 μl de una dilución 1:10 del ADNc. Los oligonucleótidos utilizados para amplificar las histonas fueron diseñados en la región 3’ UTR de cada gen para evitar reacción cruzada entre ellas: H2A1 (qRT-H2A1 F - qRT-H2A1 R), H2AX (qRT-H2AX F- qRT-H2AX R) y H2AZ (qRT-H2AZ F- qRT-H2AZ R). Como control interno se utilizó β-tubulina (qRT-TUB 91 )#$& * F- qRT-TUB R). Como control de la eficiencia de diferenciación se analizaron los niveles de bag1 (qRT-BAG1 F- qRT-BAG1 R) y ldh2 (qRT-LDH2 F- qRT-LDH2 R) que se expresan específicamente en bradizoito y sag1 (qRT-SAG1 F- qRT-SAG1 R) que es un gen específico de taquizoito. La secuencia de cada uno de los oligonucleótidos se encuentran en la tabla 1. Todas las reacciones se realizaron por triplicado en el ciclador 7500 Real-time PCR system y los datos se analizaron con el programa SDS software versión 1.2.1 (Applied Biosystems, CA). Los niveles de ARNm de cada gen (medidos como CT) fueron normalizados a los niveles de β-tubulina y calibrados con respecto a los niveles en taquizoitos (2-ΔΔCT). Se realizó una curva de disociación para cada par de oligonucleótidos, confirmando que éstos amplificaban un único producto. Además, las reacciones de PCR se visualizaron en gel de agarosa 3% corroborando la presencia de un único amplicon. Se graficó el log2(2-ΔΔCT) para cada uno de los genes amplificados. 6.6. Expresión y purificación de las histonas recombinantes La secuencia codificante de cada una de las histonas H2A y H2B fueron liberadas del vector de clonado (pGEM-T) en el cual se encuentran con las enzimas de restricción incluidas en los oligonucleótidos específicos. Las CDSs de H2Bv y H2AZ se clonaron en los sitios BamHI/KpnI y BamHI/HindIII del plásmido pQE30 (Qiagen), respectivamente y se transformaron bacterias E. coli M15. Mientras que las CDSs de H2Ba, H2AX y H2A1 fueron clonadas entre los sitios BamHI y KpnI del vector de expresión PRSET-A (Invitrogen Life Technologies), con los cuales se transformaron E. coli BL21pLys. Ambos plásmidos permiten la expresión de proteínas recombinantes fusionadas a una cola de poli-histidinas en su región N-terminal. Las histonas se expresaron como proteínas recombinantes mediante el cultivo bacteriano en medio líquido LB (Luria-Bertoni). Cuando el crecimiento presentó OD(λ=600) = 0,6, se realizó la inducción con IPTG (0,1 mM) en agitación a 30°C toda la noche (o.n.). Las proteínas fueron purificadas utilizando columna de niquel (QIAGEN) siguiendo las instrucciones del fabricante. Por otro lado, se amplificaron por PCR los primeros 120 nucleótidos de la CDS, correspondientes a la región N-terminal de H2AZ (H2AZ-Nt). Se utilizó el oligonucleótido sentido H2AZ F (Tabla 1) y el antisentido H2AZ-Nt (5’-GTCGACAAGAGGAGCCTTCTTGCC). La secuencia resultante fue clonada en los sitios BamHI y SalI del vector pGEX-4T-1 (Invitrogen Life Technologies), río abajo y en fase con la proteína GST (glutatión Stransferasa) de Schistosoma japonicum. La proteína recombinante se expresó en E. coli BL21pLys del mismo modo antes descripto. Luego fue purificada con una columna de Glutatión-agarosa (Amersham bioscience) como indican las instrucciones del fabricante. 92 )#$& * 6.7. Producción de anticuerpos Con el objeto de obtener anticuerpos específicos para cada una de las histonas H2A y H2B, se inmunizaron conejos y ratones con las proteínas recombinantes. Las correspondientes a H2Bv, H2AZ y H2AX (rH2Bv, rH2AZ y rH2AX respectivamente) fueron utilizadas para inmunizar conejos, mientras que rH2Ba, rH2A1 y rH2AZ-Nt fueron utilizadas para inmunizar ratones. En todos los casos se tomaron muestras de sangre de los animales previo a su inmunización, con el fin de obtener los sueros preinmunes. Los conejos fueron inmunizados con 300 μg de cada proteína recombinante emulsionada con adyuvante de Freund completo (Sigma). Tres dosis adicionales fueron administradas cada 15 días con la misma cantidad de proteína recombinante y adyuvante de Freund incompleto (Sigma). Los ratones fueron inmunizados con 10 μg de cada proteína recombinante emulsionada con adyuvante de Freund completo (Sigma). Tres dosis adicionales fueron administradas cada 15 días con la misma cantidad de proteína recombinante y adyuvante de Freund incompleto (Sigma). Para el caso de H2AZ-Nt se utilizaron 100 μg, lo cual es un estimativo correspondiente a 10 μg del péptido, teniendo en cuenta su tamaño en relación al tamaño de la GST. Para el caso de H2A1 se obtuvieron dos lotes de anticuerpo: anti-H2A1 L1 (αH2A1 L1) altamente específico y anti-H2A1 L2 (α-H2A1 L2), producto de mayor número de inmunizaciones, el cual presenta reacción cruzada con H2AX. 6.8. Purificación de histonas Se colectaron taquizoitos frescos de un T-75 y se lisaron con en una solución que contenía: Hepes 20 mM, NaCl 100 mM, NaF 50 mM, EDTA 5 mM, Triton X-100 1%, deoxicolato de sodio 1 % e inhibidores de proteasas, por 10 min en hielo. Se centrifugaron a 10.000 x g por 10 min. El precipitado se resuspendió en 1 ml de agua y se agregaron luego 22 μl de H2SO4 concentrado y se incubaron como mínimo 1 hora en hielo para extraer en forma ácida las histonas. Estas se precipitaron con 10 volúmenes de acetona y resuspendieron en 60 μl de solución de siembra correspondiente a los geles a utilizar. 93 )#$& * 6.9. Análisis de histonas purificadas por TAU-PAGE y MALDITOF-TOF. Las histonas purificadas fueron separadas por electroforesis en gel de poliacrilamida conteniendo Triton, ácido acético y urea (TAU-PAGE). Esta técnica separa proteínas por carga y masa; lo cual permite diferenciar proteínas con pesos moleculares muy similares y/o modificaciones postraduccionales de las mismas. El gel separador contiene: acrilamida 12 %, bis-acrilamida 0,32 %, urea 8 M y Triton X-100 10 mM. El gel de apilamiento contiene: acrilamida 3,9 %, bis-acrilamida 0,2 % y urea 8 M. La polimerización de los mismos se llevó a cabo con riboflavina y TEMED en presencia de luz. Las histonas extraídas de taquizoitos crecidos en un frasco T-75 se resuspendieron en solución de siembra TAU-PAGE (urea 8 M, β-mercaptoetanol 5 %, ácido acético glacial 6 % y una pizca de Pyronin Y que es un colorante ácido rosado) y se separaron por TAU-PAGE utilizando como tampón de corrida Glicina 0,1 M y ácido acético glacial 1 M. El gel resultante fue teñido con Coomassie blue y teniendo en cuenta los cuidados necesarios al realizar espectrometría de masa, se seleccionaron y cortaron diferentes bandas. Estas fueron digeridas con tripsina y enviadas al laboratorio de Ulf Hellman (Uppsala University, Suecia) donde fueron analizadas por “ Matrix Assisted Laser Desorption Ionization Time of Flight Mass Spectrometry” (MALDI-ToF-MS) utilizando Ultraflex TOF/TOF (Bruker Daltonics, Bremen, Germany). La identificación de las proteínas se realizó con el programa “ProFound search engine” y la base de datos NCBInr. Cuando se determinó necesario o importante, ciertos péptidos fueron secuenciados por Post Source Decay (PSD) (Hellman and Bhikhabhai 2002). Las histonas separadas por TAU-PAGE, también fueron analizadas por ensayos de Western blot (ver a continuación). En este caso los geles fueron lavados repetidas veces con agua y luego transferidos como si fueran geles de SDS-PAGE. 6.10. Análisis de Western Blot Se utilizaron ensayos de Western blot (WB) para analizar proteínas recombinantes, extractos proteicos de taquizoitos, bradizoitos o células HFF (control negativo) e histonas purificadas. Las muestras fueron preparadas de la siguiente manera: Proteínas recombinantes: se cuantificaron por Bradford o absorbancia a λ=280 nm y se resuspendieron en solución de siembra SDS-PAGE en una concentración final de 1 μg/10 μl. 94 )#$& * Parásitos y células: se colectaron y centrifugaron por 10 min a 2000 rpm. Se lavaron con PBS y se resuspendieron 1x107 parásitos cada 20 μl de solución de siembra SDS-PAGE. Las muestras se hirvieron por 10 min. Histonas purificadas: las histonas purificadas por extracción ácida de taquizoitos provenientes de un T-75, se resuspendieron en 60 μl de solución de siembra SDS-PAGE. Para ello se utilizó una solución de siembra SDS-PAGE 5x; la cual contiene TrisHCl (pH 6,8) 60 mM, glicerol 25%, SDS 2%, 2β-mercapto etanol 2 ml y azul de bromofenol 0,1%. Veinte microlitros de las muestras así preparadas fueron sembradas por calle y separadas por electroforesis en geles de poliacrilamida conteniendo SDS (SDS-PAGE) bajo condiciones reductoras, según se describe en (Sambrook and Russell 2001). Luego fueron transferidas a membranas de nitrocelulosa o PVDF (poliviniliedeno fluoruro). Previo a iniciar el ensayo de WB, las membranas fueron teñidas con rojo Ponceau para corroborar la correcta transferencia de las proteínas. Luego se bloquearon los sitios de pegado inespecífico incubando las membranas en solución tampón Tris-salino (TBS: 100 mM Tris-HCl, 0,9% NaCl, pH 7,5) suplementado con 0,05% Tween-20 y leche descremada 5% (TBS-T-L). Posteriormente, las membranas fueron incubadas 1 h a temperatura ambiente con el anticuerpo policlonal específico diluido en TBS-T-L a las diluciones establecidas en la Tabla 2. Después de que las membranas fueron lavadas con TBS-T tres veces por 5 min, se incubaron con los correspondientes anticuerpos secundarios diluido en TBS-T-L por 1 h a temperatura ambiente. A continuación se lavaron con TBS-T tres veces por 5 min, procediendo finalmente con el revelado del mismo. Dos sistemas diferentes fueron utilizados: uno en el que se utilizaron anticuerpos secundarios de cabra anti-ratón/conejo conjugados con fosfatasa alcalina (Sigma; 1/30.000) y revelados con NBT-BCIP (Promega). Y el otro, en el que los anticuerpos de cabra anti-ratón/conejo se encontraban conjugados con peroxidasa de rabanito (1/4.000), que fueron revelados con el sistema de detección ECL (Amersham-GE). 95 )#$& * Tabla 2. Anticuerpos utilizados en WB, IFI e IP. Anticuerpo (anti-) Origen Dilución WB Dilución IFI H2Bv Conejo, generado en el laboratorio 1/5.000 1/500 H2A1 L1 Ratón, generado en el laboratorio 1/300 (o.n.) 1/50 H2A1 L2 Ratón, generado en el laboratorio 1/300 (o.n.) 1/50 H2AX Conejo, generado en el laboratorio 1/5.000 1/500 H2AZ Conejo, generado en el laboratorio 1/10.000 1/500 H2AZ-Nt Ratón, generado en el laboratorio 1/5.000 1/500 tubulina Conejo, donado por Dr. David Sibley (Washington University, MO) 1/3.000 actina Ratón, donado por Dr. 1/500 SAG1 Ratón, donado por Dr. Dubremetz (Université de Montpellier II, France) 1/3.000 1/500 BAG1 Conejo, generado en el laboratorio 1/1.000 1/300 DBL Conjugado con FICT, γH2AX Conejo, Upstate 05-636 1/4000 AcH3 Conejo, Upstate 17-615 1/1.000 AcLys Conejo, ImmuneChem ICP0380 1/1.000 c-myc Ratón, Santa Cruz Biotechnology 9E10 1/1.000 H4K20me1 Conejo, Abcam ab9051 1/1.000 1/100 1/300 6.11. Inmunofluorescencia indirecta (IFI) Células HFF en monocapa crecidas sobre cubreobjetos fueron infectadas con taquizoitos. Se cultivaron en condiciones estándar de taquizoitos por 24 horas o bajo condiciones de bradizoitos por 4 días. Luego, las células conteniendo taquizoitos o bradizoitos fueron fijadas con paraformaldehido 4% durante 30 min. A continuación, se 96 )#$& * las permeabilizó con 0,2% Tritón X-100 en PBS por 10 min y se bloquearon con albúmina sérica bovina (BSA) 1% por 1 h. Se las incubó con la dilución conveniente del anticuerpo primario específico en BSA 0,5% (Tabla 2) por 1 hora. Después de varios lavados con PBS, se incubaron con el correspondiente anticuerpo secundario Alexa fluor generados en cabra anti-ratón 594 o anti-conejo 488 (1/2000, Invitrogen Life Technologies). También se utilizaron los anticuerpos anti-ratón/conejo conjugados con isotiocianato de rodamina (TRITC) o con isotiocianato de fluoresceína (FITC) (1/200, Jackson ImmunoResearchLaboratories, Inc.). Luego de varios lavados con PBS, se incubaron con Hoescht 33342 o DAPI (4,6-diamidino 2-fenil indol) por 2 min para marcar los núcleos. Se lavaron nuevamente y se montaron en medio de montaje Fluoromont G (Southern Biotechnology Associates). Los preparados fueron analizados por microscopía de epifluorescencia (Nikon Eclipse E600). Los colores emergentes de cada filtro fueron registrados separadamente y luego mezclados usando el programa Adobe Photoshop 7.0 y/o Image Pro-plus 5.1. 6.12. Determinación de la especificidad de los anticuerpos Una vez obtenidos los anticuerpos, se evaluó la especificidad y presencia de reacción cruzada para cada uno de ellos a través de dos métodos diferentes: i. Western blot, utilizando las proteínas recombinantes y ii. ELISAs competitivos. En el primer caso, 0,15 μg de las distintas proteínas recombinantes se analizaron por SDS-PAGE 15% y se transfirieron a membranas de nitrocelulosa. En todos los casos también se utilizó rROP2 como control negativo, siendo ésta una proteína recombinante no relacionada que contiene la cola de poli-histidina. Los Western blots fueron realizados como se describió previamente. Sin embargo, en cada caso el anticuerpo primario fue previamente incubado con rROP2 (50 μg/ml) por una hora a 4°C, bloqueando los posibles anticuerpos generados anti-poli-histidinas. En ningún caso hubo reacción del anti-suero con rROP2, cuando los Western blots se realizaron de este modo. Los ELISAs competitivos se realizaron de la siguiente manera. Las proteínas recombinantes rH2AZ, rH2Bv, rH2AX y rH2A1 fueron inmovilizadas en una placa de microtitulación (Immuno Plate Maxisorp; Nunc). Para ello cada pocillo fue cubierto con 100 μl de estas proteínas recombinantes diluidas a 1 μg/ml en tampón carbonato (0,05M, pH 9,7) a 4 °C por toda la noche. Al día siguiente, los pocillos fueron lavados tres veces con PBS–0.25% Tween-20 (PBS-T) y bloqueados con 200 μl de solución de bloqueo (PBS-T con 5% de leche) por 1 h a 37 °C. Luego, fueron incubados por 1 h a 37 °C con 100 μl de cada anticuerpo a ser analizado diluido en solución de bloqueo: α-H2AZ 97 )#$& * (1/5.000), α-H2Bv (1/10.000), α-H2AX (1/30.000) y α-H2A1 L2 (1/150). Cada uno de los cuales fue preincubado por 1 h a 4 °C con concentraciones crecientes de proteínas recombinantes: la proteína específica para ese anticuerpo, las proteínas que se desean evaluar si son reconocidas por reacción cruzada y una proteína no relacionada, que en todos los casos fue rROP2. A continuación se lavaron los pocillos con PBS-T y se incubaron 30 min a 37 °C con el anticuerpo secundario conjugados con proxidasa (anticonejo o ratón SIGMA, según correspondía). Los inmunocomplejos fueron lavados con PBS-T y revelados con orto-fenil etilen diamina (OPD) y peróxido de hidrógeno. Luego de 20 min, la reacción de color fue parada mediante el agregado de ácido sulfúrico 0,2 N. La absorbancia fue medida a 492 nm con un lector de microplacas automático (Rayto, RT21000). Cada determinación se realizó por duplicado y al menos dos ELISAs se llevaron a cabo de forma independiente para cada antisuero. 6.13. Coinmunoprecipitación Las inmunoprecipitaciones fueron realizadas con 5x108 taquizoitos (RHΔuprt y/o RHΔhxgprt) los cuales fueron lisados utilizando dos métodos diferentes: sonicación y digestión con endonucleasa micrococcal de Staphylococcus aureus (MNasa – Sigma N3755). En el primer caso se obtienen fragmentos de cromatina que contienen entre 100 a 600 pb, mientras que en el segundo, una solución de mononucleosomas. Los parásitos se sonicaron a 30% de amplitud por 15 s, tres veces en solución de lisis (Tris-HCl (pH8) 50mM, NaCl 150 mM, EDTA 4 mM, NP-40 1%, butirato de sodio 20mM como inhibidor de acetilasas, más inhibidor de proteasas). Se centrifugaron 10 min a 10.000 x g y el sobrenadante fue utilizado para realizar la inmunoprecipitación. El 10% fue sometido a extracción del ADN con fenol/cloroformo para analizar el tamaño de los fragmentos obtenidos. Luego, se precipitaron con etanol absoluto a 4 °C o.n., centrifugadas y resuspendidas en agua. Las muestras fueron visualizadas mediante electroforesis en gel de agarosa 1% y tinción con bromuro de etidio. El tratamiento con MNasa se llevó a cabo de la siguiente manera. Los parásitos fueron lavados y resuspendidos en solución de permeabilización (KCl 100 mM; Tris (pH 8.0) 10 mM; EDTA 25 mM; DTT 1 mM) y permeabilizados con digitonina (40μM) por 5 min a temperatura ambiente. Se lavaron y resuspendieron en 1 ml de solución isotónica fría (KCl 100 mM; Tris (pH 8) 10 mM; CaCl2 10 mM; glycerol 5%; DTT 1 mM; PMSF 1 mM); al cual se le agregaron 6 unidades de MNasa y se incubaron por 15 min a 28 °C. La reacción fue detenida con EGTA (concentración final de 10 mM). Para mejorar la solubilidad de la cromatina se agregó NP-40 y NaCl en una concentración final de 0,5% y 98 )#$& * 200 mM, respectivamente. Luego de centrifugar a 10.000 x g por 10 min a 4°C, el sobrenadante fue utilizado para realizar la inmunoprecipitación. La presencia de mononucleosomas fue evaluada mediante extracción del ADN de una alícuota (5%) con fenol/cloroformo, precipitación con alcohol y visualización en gel de agarosa 1,5% con bromuro de etidio. Tanto los parásitos sonicados como la solución de mononucleosomas, fueron incubados con 25 μl de los anticuerpos policlonales obtenidos en el laboratorio o 5 μl de los anticuerpos comerciales, o.n. a 4°C. Al día siguiente se agregó el doble de proteína A/G plus (Santa Cruz) y se incubó por dos horas más. Luego de 6 lavados con solución de lisis, los complejos colectados fueron resuspendidos en 60 μl de solución de siembra para proteínas y hervidas por 10 min. Veinte μl/calle de cada muestra fueron separados por SDS-PAGE y analizados por WB. Se realizaron inmunoprecipitaciones con el anticuerpo preinmune como control negativo. La ausencia de proteínas contaminantes se corroboró mediante WB con anti-SAG1. También se realizó la corrida electroforética del anticuerpo unido a la proteína A/G en paralelo con cada inmunoprecipitación para identificar las bandas correspondientes al anticuerpo. Los anticuerpos utilizados se encuentran listados en la tabla 2. 6.14. Inmunoprecipitación de cromatina (ChIP) Para conocer la localización genómica de las histonas, y por lo tanto dilucidar su rol en la modulación de la cromatina, se realizaron experimentos de inmunoprecipitación de cromatina (ChIP). Se colectaron taquizoitos RH, se filtraron y fijaron con 1% paraformaldehido por 10 min a 37°C. Se centrifugaron, lavaron con PBS y resuspendieron a 5x107 parásitos/200 μl de tampón de lisis fría (SDS 1%,EDTA 10 mM, Tris-HCl (pH 8,1) 50 mM, butirato de sodio 20 mM, más inhibidor de proteasas Sigma P8340). Todos los pasos se realizaron a 4°C a no ser que se indique lo contrario. Se incubaron por 10 min en hielo y se dividieron en 200 μl por reacción. Cada tubo fue sonicado y centrifugado a 10.000 x g por 10 min con el fin de obtener fragmentos de cromatina que contengan entre 1000 y 100 pb. Cada sobrenadante se diluyó 10 veces en tampón de dilución (SDS 0,01 %, triton X-100 1,1 %, EDTA 1,2 mM, Tris-HCl (pH 8,1) 16,7 mM, NaCl 167 mM y butirato de sodio 20 mM, más cocktail inhibidor de proteasas). Se reservó el 10 % de las muestras como cantidad total de ADN (input) presente en las mismas, el resto fue incubado con 80 μl de proteína A agarose con ADN de esperma de salmón (Upstate) por 30 min con agitación, para eliminar uniones inespecíficas. Luego de centrifugar 1 min a 1.000 rpm, el sobrenadante se incubó o.n. con: 10 μl de los 99 )#$& * anticuerpos generados en el laboratorio (α-H2AX, α-H2AZ o α-H2Bv) o 5 μl de anticuerpos comerciales (α-AcH3 o α-H4K20me1). Como control negativo se incubó un tubo con suero preinmune de H2AX. Al día siguiente se agregaron 60 μl de proteína A agarose con ADN de esperma de salmón y se incubaron por 1 hora con rotación a 4 °C. Los complejos anticuerpo-proteína-ADN fueron lavados 3 veces con 1 ml de cada una de las siguientes soluciones: “Tampón de lavado con baja concentración de sal” (SDS 0,1 %, Triton X-100 1 %, EDTA 2 mM, Tris-HCl 20 mM, NaCl 150 mM, pH 8,1); “Tampón de lavado con alta concentración de sal” (SDS 0,1 %, Triton-X 100 1 %, EDTA 2 mM, TrisHCl 20 mM, NaCl 500 mM, pH 8,1) y “Tampón de lavado con LiCl” (LiCl 0,25 M, NP-40 1 %, deoxicolato de sodio 1 %, EDTA 1 mM, Tris-HCl 10 mM, pH 8,1); y 6 veces con solución TE (Tris-HCl 10 mM, EDTA 1 mM, pH 8,0). A continuación fueron eluídas con 250 μl de Tampón de Elución (SDS 1%, NaHCO3 0,1 M) a temperatura ambiente por 15 min con agitación, por duplicado. Para revertir la fijación, tanto de los eluatos combinados como de los Input, se les agregaron 20 μl de NaCl (5M) e incubaron a 65oC o.n. Luego, fueron tratadas con proteinasa K y el ADN asociado a las histonas analizadas, fue purificado con el kit “PCR Purification Kit” (Qiagen). Estas muestras de ADN fueron analizadas por PCR en tiempo real (qPCR). Las reacciones de amplificación se llevaron a cabo en un volumen final de 15 μl conteniendo SYBR Green PCR Master Mix (Roche) 1 μl de ADN como templado y 0,25 μM de oligonucleótidos sentido y antisentido diseñados para amplificar las regiones promotoras de ldh2, β-tubulin, sag1 y bag1 y dos regiones diferentes de TgIRE. Las secuencias de los oligonucleótidos se encuentran listadas en la Tabla 3. La curva de calibración se obtuvo usando como templado 2%, 25% y 200% del Input. Todas las reacciones se llevaron a cabo por triplicado usando el ciclador Mx3005P (Stratagene) y se analizaron con el programa MxPro-Mx3005P v 4.01 Build 369 Schema 80 2007 (Stratagene). Se realizó una curva de disociación para cada par de oligonucleótidos, confirmando que éstos amplificaban un único producto. Además, las reacciones de PCR se visualizaron en gel de agarosa 3% corroborando la presencia de un único amplicon. 100 )#$& * Tabla 3. Oligonucleótidos utilizados en ChIP. Descripción Producto amplificado Nombre Secuencia q-TUB F q-TUB R 5’-TCCTTTTCTTCATGCACTCGC 5’-GTCGTTCGAATTCTCTGCCC Promotor de β-tubulina - 627 a - 526 q-BAG1 F q-BAG1 R 5’-TCCTCCCCTGGATCTTCCTCC 5’-TTGCACAAAACTGGCAAAGG Promotor de bag1 - 739 a - 638 q-LDH2 F q-LDH2 R 5’-AAGTGTGCACGCTTTGCAAG 5’-CATGCTCCGGGCAGTACCT Promotor de ldh2 - 505 a - 404 q-TgIRE 100 F q-TgIRE 100 R 5’-TTCTTCACGGTTCCGGACTT 5’-CAGCAGTAGCCAACCACGT TgIRE 3 a 96 q-TgIRE 600 F q-TgIRE 600 R 5’-GTTATGAGTCCACCACACGGG 5’-CATGATGTGCATGAAACGTGC TgIRE 732 a 825 q-SAG1 F q-SAG1 R 5’-AACTTCCCACACGAGGCATT 5’-GATCGGCCCCTAATTCAGC Promotor de sag1 (pb desde el ATG) - 653 a - 560 q: utilizados para qPCR. F: sentido (del inglés Forward). R: antisentido (del inglés Reverse). 6.15. Tratamiento con peróxido de hidrógeno Taquizoitos frescos y filtrados fueron tratados con 0, 100, 200 o 400 μM de peróxido de hidrógeno (H2O2) por 1 h a 37oC con CO2 5 %. Luego los parásitos fueron colectados a 2.000 x g por 5 min y lavados con PBS. Cada muestra se dividió en dos. A una fracción se les extrajo rápidamente el ARN, del cual se obtuvo ADNc por transcripción reversa, como se describe en la sección 6.5 - H2As. Se analizó la expresión de los genes de las histonas por qRT-PCR. Todas las reacciones se llevaron a cabo por triplicado usando el ciclador 7500 Real-time PCR system y se analizaron con el programa SDS software versión 1.2.1 (Applied Biosystems, CA). Los niveles de ARNm de cada gen (medidos como CT) fueron normalizados a los niveles de β -tubulina y calibrados con respecto a los niveles en el control de taquizoitos sin H2O2 (2-ΔΔCT). Se realizó una curva de disociación para cada par de oligonucleótidos, confirmando que éstos amplificaban un único producto. Además, las reacciones de PCR se visualizaron en gel de agarosa 3% corroborando la presencia de un único amplicon. Se graficó el log2(2-ΔΔCT) para cada uno de los genes amplificados. La otra fracción fue analizada por WB. Para ello, los parásitos fueron sonicados y los extractos separados en 4-12 % NuPAGE minigels con Tampón de corrida MES (Invitrogen Life Technologies). Luego fueron transferidos y analizados por WB utilizando anti-H2AX (α-H2AX), anti-H2AX fosforilada (α-γH2AX) y anti-tubulina como control de 101 )#$& * carga (Tabla 2). Los Western blots fueron revelados con anticuerpos de cabra antiratón/conejo conjugados con peroxidasa de rabanito (1/4.000) y revelados con el sistema de detección ECL (Amersham-GE). La intensidad de las bandas fueron cuantificadas utilizando el programa Gel Pro Analyzer version 4.0 (Media Cybernetics) utilizando el análisis “gel 1D” y determinando la densidad óptica integrada (IOD). 6.16. Construcción de plásmidos de expresión en Toxoplasma Con el objetivo de obtener parásitos que sobreexpresen H2AX, se generó la construcción pTyH2AX. oligonucleótidos: 5’- Se amplificó su CDS por PCR utilizando los siguientes ATGCATATGTCCGCCAAAGGTGCAGG (sentido) y 5’- TTAATTAAAGGATAGCCATTCTGTCTGGTG (antisentido). Los sitios de restricción Nsi I y Pac I fueron incluidos en los oligonucleótidos sentido y antisentido respectivamente (secuencia subrayada). El fragmento obtenido se clonó en el vector pGEM-T easy vector (promega) y se secuenció. A continuación se lo digirió con Nsi I y Pac I y se purificó el inserto, que fue introducido en el plásmido pTUB8MycGFPPf.myo tailTy-1/HX (cedido gentilmente por la Dra. D. Soldati, Université de Genéve, Suiza), bajo el promotor de tubulina de T. gondii y río abajo y en fase con la secuencia que codifica para c-myc. Este vector contiene cassettes de resistencia a ampicilina (para el clonado en bacterias) y el gen de selección hxgprt (permite la selección de parásitos RHΔhxgprt transfectados). 6.17. Transfección de parásitos La construcción pTyH2AX fue utilizada para transfectar por electroporación parásitos RHΔhxgprt. La electroporación se llevó a cabo como describió Donald et al (Donald et al. 1996) . Brevemente, se electroporaron 2x107 taquizoitos y 50 μg de la construcción, todo resuspendido en CYTOMIX (120 mM KCl; 0,15mM CaCl2; 10 mM K2HPO4/KH2PO4 pH: 7,6; 25 mM HEPES pH: 7,6; 2 mM EDTA pH: 7,6; 5 mM MgCl2; 3 mM ATP; 3 mM Glutatión), en cubetas de 2 mm (BTX). El electroporador empleado fue BTX EMC 630 con las siguientes condiciones: volts 1,5 kv y resistencia 25 Ω. Los parásitos electroporados fueron cultivados por tres pasajes utilizando 25 μg/ml de ácido micofenólico y 50 μg/ml de xantina como drogas de selección. El ácido micofenólico es un potente inhibidor de la enzima IMP deshidrogenasa y por lo tanto bloquea la síntesis de novo de guanina a partir de AMP. Mediante la adición de xantina en el medio de cultivo, solo los parásitos que hayan incorporado el plásmido, conteniendo el gen de selección hxgprt, serán capaces de sintetizar guanina a partir de xantina (Figura 6.18.1). Una vez 102 )#$& * obtenida la población estable, se los clonó por dilución múltiple y se aislaron varios clones. Estos fueron analizados por IFI y WB utilizando α-c-myc y α-H2AX (Tabla 2). Adenina AMP Adenosina Inosina Hipoxantina hxgprt IMP hxgprt Hipoxantina MPA hxgprt XMP GMP Xantina Figura 6.18.1. Vía de síntesis de guanosina monofosfato (GMP) y selección HX. Se utilizan parásitos KO para hxgprt, los cuales son capaces de sintetizar GMP a través de la interconversión entre adenosina monofosfato (AMP) y inosina monofosfato (IMP). Al agregarse ácido micofenólico (MPA) se bloquea este paso, por lo que sólo los parásitos que hayan incorportado el plásmido que contiene hxgprt serán capaces de sintetizar GMP a través de la Xantina agregada al medio. 6.18. Ensayo de replicación. Se utilizaron para comparar el crecimiento de los parásitos que sobreexprersan H2AX (mycH2AX, clones estables para pTyH2AX) con respecto a los parásitos RHΔhxgprt. Se infectaron células HFF crecidas en monocapa sobre cubreobjetos con 8 x 105 parásitos por duplicado. Se indujo la invasión de los mismos incubándolos por 10 min en hielo y luego 1 h a 37°C con CO2 5 %. A continuación se lavaron con PBS extensivamente para eliminar los parásitos extracelulares y se incubaron en condiciones estándar de taquizoitos por 16 horas. Las células fueron fijadas con paraformaldehido 4% durante 30 min y montados con Fluoromont G (Southern Biotechnology Associates). El número de taquizoitos por vacuola fue contado utilizando el objetivo de 100x. Se contaron 100 vacuolas por duplicado, por muestra. Para realizar el análisis estadístico de los datos, se determinó el número total de parásitos correspondiente a las 100 vacuolas para cada caso y se aplicó una Anova simple empleando el método de Holm-Sidak. Los gráficos y análisis estadísticos se realizaron con el programa Sigma Stat v3.5 acoplado al Sigma Plot v10.0 (Systat Software, Inc.). 103 ,$ Bibliografía Adam, M., F. Robert, M. Larochelle, and L. Gaudreau. 2001. H2A.Z is required for global chromatin integrity and for recruitment of RNA polymerase II under specific conditions. Mol Cell Biol 21: 6270-9. Ahn, S. H., K. A. Henderson, S. Keeney, and C. D. Allis. 2005. H2B (Ser10) phosphorylation is induced during apoptosis and meiosis in S. cerevisiae. Cell Cycle 4: 780-3. Ahn, S. H., R. L. Diaz, M. Grunstein, and C. D. Allis. 2006. Histone H2B deacetylation at lysine 11 is required for yeast apoptosis induced by phosphorylation of H2B at serine 10. Mol Cell 24: 211-20. Albig, W., R. Trappe, E. Kardalinou, S. Eick, and D. Doenecke. 1999. The human H2A and H2B histone gene complement. Biol Chem 380: 7-18. Alsford, S., and D. Horn. 2004. Trypanosomatid histones. Mol Microbiol 53: 365-72. Barski, A., S. Cuddapah, K. Cui, T. Y. Roh, D. E. Schones, Z. Wang, G. Wei, I. Chepelev, and K. Zhao. 2007. High-resolution profiling of histone methylations in the human genome. Cell 129: 823-37. Bassing, C. H., H. Suh, D. O. Ferguson, K. F. Chua, J. Manis, M. Eckersdorff, M. Gleason, R. Bronson, C. Lee, and F. W. Alt. 2003. Histone H2AX: a dosagedependent suppressor of oncogenic translocations and tumors. Cell 114: 359-70. Bassing, C. H., K. F. Chua, J. Sekiguchi, H. Suh, S. R. Whitlow, J. C. Fleming, B. C. Monroe, D. N. Ciccone, C. Yan, K. Vlasakova, D. M. Livingston, D. O. Ferguson, R. Scully, and F. W. Alt. 2002. Increased ionizing radiation sensitivity and genomic instability in the absence of histone H2AX. Proc Natl Acad Sci U S A 99: 8173-8. Behnke, M. S., J. B. Radke, A. T. Smith, W. J. Sullivan, Jr., and M. W. White. 2008. The transcription of bradyzoite genes in Toxoplasma gondii is controlled by autonomous promoter elements. Mol Microbiol 68: 1502-18. Berger, S. L. 2002. Histone modifications in transcriptional regulation. Curr Opin Genet Dev 12: 142-8. Bewersdorf, J., B. T. Bennett, and K. L. Knight. 2006. H2AX chromatin structures and their response to DNA damage revealed by 4Pi microscopy. Proc Natl Acad Sci U S A 103: 18137-42. Bohne, W., and D. S. Roos. 1997. Stage-specific expression of a selectable marker in Toxoplasma gondii permits selective inhibition of either tachyzoites or bradyzoites. Mol Biochem Parasitol 88: 115-26. Boothroyd, J. C., A. Hehl, L. J. Knoll, and I. D. Manger. 1998. The surface of Toxoplasma: more and less. Int J Parasitol 28: 3-9. Boulard, M., T. Gautier, G. O. Mbele, V. Gerson, A. Hamiche, D. Angelov, P. Bouvet, and S. Dimitrov. 2006. The NH2 tail of the novel histone variant H2BFWT exhibits properties distinct from conventional H2B with respect to the assembly of mitotic chromosomes. Mol Cell Biol 26: 1518-26. Buschbeck, M., I. Uribesalgo, I. Wibowo, P. Rue, D. Martin, A. Gutierrez, L. Morey, R. Guigo, H. Lopez-Schier, and L. Di Croce. 2009. The histone variant macroH2A is an epigenetic regulator of key developmental genes. Nat Struct Mol Biol 16: 1074-9. Carr, A. M., S. M. Dorrington, J. Hindley, G. A. Phear, S. J. Aves, and P. Nurse. 1994. Analysis of a histone H2A variant from fission yeast: evidence for a role in chromosome stability. Mol Gen Genet 245: 628-35. Celeste, A., S. Petersen, P. J. Romanienko, O. Fernandez-Capetillo, H. T. Chen, O. A. Sedelnikova, B. Reina-San-Martin, V. Coppola, E. Meffre, M. J. 104 ,$ Difilippantonio, C. Redon, D. R. Pilch, A. Olaru, M. Eckhaus, R. D. CameriniOtero, L. Tessarollo, F. Livak, K. Manova, W. M. Bonner, M. C. Nussenzweig, and A. Nussenzweig. 2002. Genomic instability in mice lacking histone H2AX. Science 296: 922-7. Clarkson, M. J., J. R. Wells, F. Gibson, R. Saint, and D. J. Tremethick. 1999. Regions of variant histone His2AvD required for Drosophila development. Nature 399: 6947. Clemente, M., N. de Miguel, V. V. Lia, M. Matrajt, and S. O. Angel. 2004. Structure analysis of two Toxoplasma gondii and Neospora caninum satellite DNA families and evolution of their common monomeric sequence. J Mol Evol 58: 557-67. Chadwick, B. P., and H. F. Willard. 2001. A novel chromatin protein, distantly related to histone H2A, is largely excluded from the inactive X chromosome. J Cell Biol 152: 375-84. Chen, H. T., A. Bhandoola, M. J. Difilippantonio, J. Zhu, M. J. Brown, X. Tai, E. P. Rogakou, T. M. Brotz, W. M. Bonner, T. Ried, and A. Nussenzweig. 2000. Response to RAG-mediated VDJ cleavage by NBS1 and gamma-H2AX. Science 290: 1962-5. Daniel, J. A., M. S. Torok, Z. W. Sun, D. Schieltz, C. D. Allis, J. R. Yates, 3rd, and P. A. Grant. 2004. Deubiquitination of histone H2B by a yeast acetyltransferase complex regulates transcription. J Biol Chem 279: 1867-71. de la Barre, A. E., D. Angelov, A. Molla, and S. Dimitrov. 2001. The N-terminus of histone H2B, but not that of histone H3 or its phosphorylation, is essential for chromosome condensation. EMBO J 20: 6383-93. de la Barre, A. E., V. Gerson, S. Gout, M. Creaven, C. D. Allis, and S. Dimitrov. 2000. Core histone N-termini play an essential role in mitotic chromosome condensation. EMBO J 19: 379-91. Dickey, J. S., C. E. Redon, A. J. Nakamura, B. J. Baird, O. A. Sedelnikova, and W. M. Bonner. 2009. H2AX: functional roles and potential applications. Chromosoma. Donald, R. G., D. Carter, B. Ullman, and D. S. Roos. 1996. Insertional tagging, cloning, and expression of the Toxoplasma gondii hypoxanthine-xanthine-guanine phosphoribosyltransferase gene. Use as a selectable marker for stable transformation. J Biol Chem 271: 14010-9. Downs, J. A., N. F. Lowndes, and S. P. Jackson. 2000. A role for Saccharomyces cerevisiae histone H2A in DNA repair. Nature 408: 1001-4. Durlach, R., F. Kaufer, L. Carral, C. Freuler, M. Ceriotto, M. Rodriguez, H. Freilij, J. Altcheh, L. Vazquez, R. Corazza, M. Dalla Fontana, H. Arienti, E. Sturba, S. Gonzalez Ayala, E. Cecchini, C. Salomon, M. Nadal, N. Gutierrez, and E. Guarnera. 2008. [Argentine Consensus of Congenital Toxoplasmosis]. Medicina (B Aires) 68: 75-87. Echeverria, P. C., P. A. Rojas, V. Martin, E. A. Guarnera, V. Pszenny, and S. O. Angel. 2000. Characterisation of a novel interspersed Toxoplasma gondii DNA repeat with potential uses for PCR diagnosis and PCR-RFLP analysis. FEMS Microbiol Lett 184: 23-7. Echeverria, P. C., M. Matrajt, O. S. Harb, M. P. Zappia, M. A. Costas, D. S. Roos, J. F. Dubremetz, and S. O. Angel. 2005. Toxoplasma gondii Hsp90 is a potential drug target whose expression and subcellular localization are developmentally regulated. J Mol Biol 350: 723-34. Escargueil, A. E., D. G. Soares, M. Salvador, A. K. Larsen, and J. A. Henriques. 2008. What histone code for DNA repair? Mutat Res 658: 259-70. Faast, R., V. Thonglairoam, T. C. Schulz, J. Beall, J. R. Wells, H. Taylor, K. Matthaei, P. D. Rathjen, D. J. Tremethick, and I. Lyons. 2001. Histone variant H2A.Z is required for early mammalian development. Curr Biol 11: 1183-7. Ferguson, D. J., W. M. Hutchison, J. F. Dunachie, and J. C. Siim. 1974. Ultrastructural study of early stages of asexual multiplication and microgametogony of 105 ,$ Toxoplasma gondii in the small intestine of the cat. Acta Pathol Microbiol Scand B Microbiol Immunol 82: 167-81. Fernandez-Capetillo, O., A. Lee, M. Nussenzweig, and A. Nussenzweig. 2004. H2AX: the histone guardian of the genome. DNA Repair (Amst) 3: 959-67. Fernandez-Capetillo, O., S. K. Mahadevaiah, A. Celeste, P. J. Romanienko, R. D. Camerini-Otero, W. M. Bonner, K. Manova, P. Burgoyne, and A. Nussenzweig. 2003. H2AX is required for chromatin remodeling and inactivation of sex chromosomes in male mouse meiosis. Dev Cell 4: 497-508. Fernandez-Capetillo, O., H. T. Chen, A. Celeste, I. Ward, P. J. Romanienko, J. C. Morales, K. Naka, Z. Xia, R. D. Camerini-Otero, N. Motoyama, P. B. Carpenter, W. M. Bonner, J. Chen, and A. Nussenzweig. 2002. DNA damage-induced G2M checkpoint activation by histone H2AX and 53BP1. Nat Cell Biol 4: 993-7. Figueiredo, L. M., L. A. Pirrit, and A. Scherf. 2000. Genomic organisation and chromatin structure of Plasmodium falciparum chromosome ends. Mol Biochem Parasitol 106: 169-74. Figueiredo, L. M., L. H. Freitas-Junior, E. Bottius, J. C. Olivo-Marin, and A. Scherf. 2002. A central role for Plasmodium falciparum subtelomeric regions in spatial positioning and telomere length regulation. EMBO J 21: 815-24. Fischle, W., Y. Wang, and C. D. Allis. 2003. Histone and chromatin cross-talk. Curr Opin Cell Biol 15: 172-83. Frenkel, J. K. 1967. Adoptive immunity to intracellular infection. J Immunol 98: 1309-19. Garcia-Salcedo, J. A., P. Gijon, and E. Pays. 1999. Regulated transcription of the histone H2B genes of Trypanosoma brucei. Eur J Biochem 264: 717-23. Gautier, T., D. W. Abbott, A. Molla, A. Verdel, J. Ausio, and S. Dimitrov. 2004. Histone variant H2ABbd confers lower stability to the nucleosome. EMBO Rep 5: 715-20. Gissot, M., K. A. Kelly, J. W. Ajioka, J. M. Greally, and K. Kim. 2007. Epigenomic modifications predict active promoters and gene structure in Toxoplasma gondii. PLoS Pathog 3: e77. Gottesfeld, J. M., J. M. Belitsky, C. Melander, P. B. Dervan, and K. Luger. 2002. Blocking transcription through a nucleosome with synthetic DNA ligands. J Mol Biol 321: 249-63. Grigg, M. E., and Y. Suzuki. 2003. Sexual recombination and clonal evolution of virulence in Toxoplasma. Microbes Infect 5: 685-90. Grove, G. W., and A. Zweidler. 1984. Regulation of nucleosomal core histone variant levels in differentiating murine erythroleukemia cells. Biochemistry 23: 4436-43. Guillemette, B., A. R. Bataille, N. Gevry, M. Adam, M. Blanchette, F. Robert, and L. Gaudreau. 2005. Variant histone H2A.Z is globally localized to the promoters of inactive yeast genes and regulates nucleosome positioning. PLoS Biol 3: e384. Hake, S. B., and C. D. Allis. 2006. Histone H3 variants and their potential role in indexing mammalian genomes: the "H3 barcode hypothesis". Proc Natl Acad Sci U S A 103: 6428-35. Harris, M. E., R. Bohni, M. H. Schneiderman, L. Ramamurthy, D. Schumperli, and W. F. Marzluff. 1991. Regulation of histone mRNA in the unperturbed cell cycle: evidence suggesting control at two posttranscriptional steps. Mol Cell Biol 11: 2416-24. Hellman, U., and R. Bhikhabhai. 2002. Easy amino acid sequencing of sulfonated peptides using post-source decay on a matrix-assisted laser desorption/ionization time-of-flight mass spectrometer equipped with a variable voltage reflector. Rapid Commun Mass Spectrom 16: 1851-9. Henry, K. W., A. Wyce, W. S. Lo, L. J. Duggan, N. C. Emre, C. F. Kao, L. Pillus, A. Shilatifard, M. A. Osley, and S. L. Berger. 2003. Transcriptional activation via sequential histone H2B ubiquitylation and deubiquitylation, mediated by SAGAassociated Ubp8. Genes Dev 17: 2648-63. 106 ,$ Heo, K., H. Kim, S. H. Choi, J. Choi, K. Kim, J. Gu, M. R. Lieber, A. S. Yang, and W. An. 2008. FACT-mediated exchange of histone variant H2AX regulated by phosphorylation of H2AX and ADP-ribosylation of Spt16. Mol Cell 30: 86-97. Hill, D. E., S. Chirukandoth, and J. P. Dubey. 2005. Biology and epidemiology of Toxoplasma gondii in man and animals. Anim Health Res Rev 6: 41-61. Hu, K., D. S. Roos, S. O. Angel, and J. M. Murray. 2004. Variability and heritability of cell division pathways in Toxoplasma gondii. J Cell Sci 117: 5697-705. Ikura, T., S. Tashiro, A. Kakino, H. Shima, N. Jacob, R. Amunugama, K. Yoder, S. Izumi, I. Kuraoka, K. Tanaka, H. Kimura, M. Ikura, S. Nishikubo, T. Ito, A. Muto, K. Miyagawa, S. Takeda, R. Fishel, K. Igarashi, and K. Kamiya. 2007. DNA damage-dependent acetylation and ubiquitination of H2AX enhances chromatin dynamics. Mol Cell Biol 27: 7028-40. Janzen, C. J., J. P. Fernandez, H. Deng, R. Diaz, S. B. Hake, and G. A. Cross. 2006. Unusual histone modifications in Trypanosoma brucei. FEBS Lett 580: 2306-10. Jerome, M. E., J. R. Radke, W. Bohne, D. S. Roos, and M. W. White. 1998. Toxoplasma gondii bradyzoites form spontaneously during sporozoite-initiated development. Infect Immun 66: 4838-44. Kalocsay, M., N. J. Hiller, and S. Jentsch. 2009. Chromosome-wide Rad51 spreading and SUMO-H2A.Z-dependent chromosome fixation in response to a persistent DNA double-strand break. Mol Cell 33: 335-43. Kamakaka, R. T., and S. Biggins. 2005. Histone variants: deviants? Genes Dev 19: 295310. Kouzarides, T. 2007. Chromatin modifications and their function. Cell 128: 693-705. Li, A., J. M. Eirin-Lopez, and J. Ausio. 2005a. H2AX: tailoring histone H2A for chromatin-dependent genomic integrity. Biochem Cell Biol 83: 505-15. Li, B., S. G. Pattenden, D. Lee, J. Gutierrez, J. Chen, C. Seidel, J. Gerton, and J. L. Workman. 2005b. Preferential occupancy of histone variant H2AZ at inactive promoters influences local histone modifications and chromatin remodeling. Proc Natl Acad Sci U S A 102: 18385-90. Li, Y. J., X. H. Fu, D. P. Liu, and C. C. Liang. 2004. Opening the chromatin for transcription. Int J Biochem Cell Biol 36: 1411-23. Liu, X., and M. A. Gorovsky. 1996. Cloning and characterization of the major histone H2A genes completes the cloning and sequencing of known histone genes of Tetrahymena thermophila. Nucleic Acids Res 24: 3023-30. Lobo, C. A., and N. Kumar. 1999. Differential transcription of histone genes in asexual and sexual stages of Plasmodium falciparum. Int J Parasitol 29: 1447-9. Lowell, J. E., and G. A. Cross. 2004. A variant histone H3 is enriched at telomeres in Trypanosoma brucei. J Cell Sci 117: 5937-47. Lowell, J. E., F. Kaiser, C. J. Janzen, and G. A. Cross. 2005. Histone H2AZ dimerizes with a novel variant H2B and is enriched at repetitive DNA in Trypanosoma brucei. J Cell Sci 118: 5721-30. Luger, K., A. W. Mader, R. K. Richmond, D. F. Sargent, and T. J. Richmond. 1997. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature 389: 251-60. Lundin, C., M. North, K. Erixon, K. Walters, D. Jenssen, A. S. Goldman, and T. Helleday. 2005. Methyl methanesulfonate (MMS) produces heat-labile DNA damage but no detectable in vivo DNA double-strand breaks. Nucleic Acids Res 33: 3799-811. Lyons, R. E., R. McLeod, and C. W. Roberts. 2002. Toxoplasma gondii tachyzoitebradyzoite interconversion. Trends Parasitol 18: 198-201. Llinas, M., and J. L. DeRisi. 2004. Pernicious plans revealed: Plasmodium falciparum genome wide expression analysis. Curr Opin Microbiol 7: 382-7. Mahadevaiah, S. K., J. M. Turner, F. Baudat, E. P. Rogakou, P. de Boer, J. BlancoRodriguez, M. Jasin, S. Keeney, W. M. Bonner, and P. S. Burgoyne. 2001. 107 ,$ Recombinational DNA double-strand breaks in mice precede synapsis. Nat Genet 27: 271-6. Malik, H. S., and S. Henikoff. 2003. Phylogenomics of the nucleosome. Nat Struct Biol 10: 882-91. McManus, K. J., and M. J. Hendzel. 2005. ATM-dependent DNA damage-independent mitotic phosphorylation of H2AX in normally growing mammalian cells. Mol Biol Cell 16: 5013-25. Meissner, M., and D. Soldati. 2005. The transcription machinery and the molecular toolbox to control gene expression in Toxoplasma gondii and other protozoan parasites. Microbes Infect 7: 1376-84. Meneghini, M. D., M. Wu, and H. D. Madhani. 2003. Conserved histone variant H2A.Z protects euchromatin from the ectopic spread of silent heterochromatin. Cell 112: 725-36. Miao, J., Q. Fan, L. Cui, and J. Li. 2006. The malaria parasite Plasmodium falciparum histones: organization, expression, and acetylation. Gene 369: 53-65. Millar, C. B., F. Xu, K. Zhang, and M. Grunstein. 2006. Acetylation of H2AZ Lys 14 is associated with genome-wide gene activity in yeast. Genes Dev 20: 711-22. Mizuguchi, G., X. Shen, J. Landry, W. H. Wu, S. Sen, and C. Wu. 2004. ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex. Science 303: 343-8. Nathan, D., K. Ingvarsdottir, D. E. Sterner, G. R. Bylebyl, M. Dokmanovic, J. A. Dorsey, K. A. Whelan, M. Krsmanovic, W. S. Lane, P. B. Meluh, E. S. Johnson, and S. L. Berger. 2006. Histone sumoylation is a negative regulator in Saccharomyces cerevisiae and shows dynamic interplay with positive-acting histone modifications. Genes Dev 20: 966-76. Parra, M. A., and J. J. Wyrick. 2007. Regulation of gene transcription by the histone H2A N-terminal domain. Mol Cell Biol 27: 7641-8. Parra, M. A., D. Kerr, D. Fahy, D. J. Pouchnik, and J. J. Wyrick. 2006. Deciphering the roles of the histone H2B N-terminal domain in genome-wide transcription. Mol Cell Biol 26: 3842-52. Pattenden, S. G., M. J. Chandy, J. L. Gutierrez, and J. L. Workman. 2005. Chromatin dynamics rule the genome. Genome Biol 6: 355. Pehrson, J. R., and V. A. Fried. 1992. MacroH2A, a core histone containing a large nonhistone region. Science 257: 1398-400. Radke, J. R., and M. W. White. 1998. A cell cycle model for the tachyzoite of Toxoplasma gondii using the Herpes simplex virus thymidine kinase. Mol Biochem Parasitol 94: 237-47. Radke, J. R., M. N. Guerini, M. Jerome, and M. W. White. 2003. A change in the premitotic period of the cell cycle is associated with bradyzoite differentiation in Toxoplasma gondii. Mol Biochem Parasitol 131: 119-27. Radke, J. R., B. Striepen, M. N. Guerini, M. E. Jerome, D. S. Roos, and M. W. White. 2001. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Mol Biochem Parasitol 115: 165-75. Raisner, R. M., P. D. Hartley, M. D. Meneghini, M. Z. Bao, C. L. Liu, S. L. Schreiber, O. J. Rando, and H. D. Madhani. 2005. Histone variant H2A.Z marks the 5' ends of both active and inactive genes in euchromatin. Cell 123: 233-48. Rangasamy, D., L. Berven, P. Ridgway, and D. J. Tremethick. 2003. Pericentric heterochromatin becomes enriched with H2A.Z during early mammalian development. EMBO J 22: 1599-607. Rawat, D. S., I. Sharma, R. Jalah, S. Lomash, V. Kothekar, S. T. Pasha, and Y. D. Sharma. 2004. Identification, expression, modeled structure and serological characterization of Plasmodium vivax histone 2B. Gene 337: 25-35. Redon, C., D. Pilch, E. Rogakou, O. Sedelnikova, K. Newrock, and W. Bonner. 2002. Histone H2A variants H2AX and H2AZ. Curr Opin Genet Dev 12: 162-9. 108 ,$ Ren, Q., and M. A. Gorovsky. 2001. Histone H2A.Z acetylation modulates an essential charge patch. Mol Cell 7: 1329-35. Rogakou, E. P., D. R. Pilch, A. H. Orr, V. S. Ivanova, and W. M. Bonner. 1998. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem 273: 5858-68. Rogakou, E. P., W. Nieves-Neira, C. Boon, Y. Pommier, and W. M. Bonner. 2000. Initiation of DNA fragmentation during apoptosis induces phosphorylation of H2AX histone at serine 139. J Biol Chem 275: 9390-5. Saeij, J. P., J. P. Boyle, and J. C. Boothroyd. 2005. Differences among the three major strains of Toxoplasma gondii and their specific interactions with the infected host. Trends Parasitol 21: 476-81. Saksouk, N., M. M. Bhatti, S. Kieffer, A. T. Smith, K. Musset, J. Garin, W. J. Sullivan, Jr., M. F. Cesbron-Delauw, and M. A. Hakimi. 2005. Histone-modifying complexes regulate gene expression pertinent to the differentiation of the protozoan parasite Toxoplasma gondii. Mol Cell Biol 25: 10301-14. Sambrook, J., and D. W. Russell. 2001. Molecular cloning, a laboratory manual. CSHL Press. Sarcinella, E., P. C. Zuzarte, P. N. Lau, R. Draker, and P. Cheung. 2007. Monoubiquitylation of H2A.Z distinguishes its association with euchromatin or facultative heterochromatin. Mol Cell Biol 27: 6457-68. Sautel, C. F., D. Cannella, O. Bastien, S. Kieffer, D. Aldebert, J. Garin, I. Tardieux, H. Belrhali, and M. A. Hakimi. 2007. SET8-mediated methylations of histone H4 lysine 20 mark silent heterochromatic domains in apicomplexan genomes. Mol Cell Biol 27: 5711-24. Shechter, D., J. J. Nicklay, R. K. Chitta, J. Shabanowitz, D. F. Hunt, and C. D. Allis. 2009. Analysis of histones in Xenopus laevis. I. A distinct index of enriched variants and modifications exists in each cell type and is remodeled during developmental transitions. J Biol Chem 284: 1064-74. Siegel, T. N., D. R. Hekstra, L. E. Kemp, L. M. Figueiredo, J. E. Lowell, D. Fenyo, X. Wang, S. Dewell, and G. A. Cross. 2009. Four histone variants mark the boundaries of polycistronic transcription units in Trypanosoma brucei. Genes Dev 23: 1063-76. Singh, U., J. L. Brewer, and J. C. Boothroyd. 2002. Genetic analysis of tachyzoite to bradyzoite differentiation mutants in Toxoplasma gondii reveals a hierarchy of gene induction. Mol Microbiol 44: 721-33. Song, X., E. Gjoneska, Q. Ren, S. D. Taverna, C. D. Allis, and M. A. Gorovsky. 2007. Phosphorylation of the SQ H2A.X motif is required for proper meiosis and mitosis in Tetrahymena thermophila. Mol Cell Biol 27: 2648-60. Stiff, T., M. O'Driscoll, N. Rief, K. Iwabuchi, M. Lobrich, and P. A. Jeggo. 2004. ATM and DNA-PK function redundantly to phosphorylate H2AX after exposure to ionizing radiation. Cancer Res 64: 2390-6. Strahl, B. D., and C. D. Allis. 2000. The language of covalent histone modifications. Nature 403: 41-5. Sullivan, W. J., Jr. 2003. Histone H3 and H3.3 variants in the protozoan pathogens Plasmodium falciparum and Toxoplasma gondii. DNA Seq 14: 227-31. Sullivan, W. J., Jr., and M. A. Hakimi. 2006. Histone mediated gene activation in Toxoplasma gondii. Mol Biochem Parasitol 148: 109-16. Sullivan, W. J., Jr., A. Naguleswaran, and S. O. Angel. 2006. Histones and histone modifications in protozoan parasites. Cell Microbiol 8: 1850-61. Suto, R. K., M. J. Clarkson, D. J. Tremethick, and K. Luger. 2000. Crystal structure of a nucleosome core particle containing the variant histone H2A.Z. Nat Struct Biol 7: 1121-4. Tamura, K., J. Dudley, M. Nei, and S. Kumar. 2007. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol 24: 1596-9. 109 ,$ Thatcher, T. H., and M. A. Gorovsky. 1994. Phylogenetic analysis of the core histones H2A, H2B, H3, and H4. Nucleic Acids Res 22: 174-9. Trelle, M. B., A. M. Salcedo-Amaya, A. Cohen, H. G. Stunneberg, and O. N. Jensen. 2009. Global histone analysis by mass spectrometry reveals a high content of acetylated lysine residues in the malaria parasite Plasmodium falciparum. J Proteome Res. Turner, B. M. 2002. Cellular memory and the histone code. Cell 111: 285-91. van Daal, A., E. M. White, M. A. Gorovsky, and S. C. Elgin. 1988. Drosophila has a single copy of the gene encoding a highly conserved histone H2A variant of the H2A.F/Z type. Nucleic Acids Res 16: 7487-97. Van der Ploeg, L. H. 1991. Control of antigenic variation in African trypanosomes. New Biol 3: 324-30. Ward, I. M., and J. Chen. 2001. Histone H2AX is phosphorylated in an ATR-dependent manner in response to replicational stress. J Biol Chem 276: 47759-62. Weiss, L. M., and K. Kim. 2000. The development and biology of bradyzoites of Toxoplasma gondii. Front Biosci 5: D391-405. Weiss, L. M., and K. Kim [eds.]. 2007. Toxoplasma gondii. The model Apicomplexan: perspectives and methods. Academic Press, UK. Weiss, L. M., Y. F. Ma, P. M. Takvorian, H. B. Tanowitz, and M. Wittner. 1998. Bradyzoite development in Toxoplasma gondii and the hsp70 stress response. Infect Immun 66: 3295-302. Whittle, C. M., K. N. McClinic, S. Ercan, X. Zhang, R. D. Green, W. G. Kelly, and J. D. Lieb. 2008. The genomic distribution and function of histone variant HTZ-1 during C. elegans embryogenesis. PLoS Genet 4: e1000187. Wu, R. S., and W. M. Bonner. 1981. Separation of basal histone synthesis from S-phase histone synthesis in dividing cells. Cell 27: 321-30. Wyrick, J. J., and M. A. Parra. 2008. The role of histone H2A and H2B post-translational modifications in transcription: A genomic perspective. Biochim Biophys Acta. Xiao, T., C. F. Kao, N. J. Krogan, Z. W. Sun, J. F. Greenblatt, M. A. Osley, and B. D. Strahl. 2005. Histone H2B ubiquitylation is associated with elongating RNA polymerase II. Mol Cell Biol 25: 637-51. Yuan, G. C., Y. J. Liu, M. F. Dion, M. D. Slack, L. F. Wu, S. J. Altschuler, and O. J. Rando. 2005. Genome-scale identification of nucleosome positions in S. cerevisiae. Science 309: 626-30. Zalensky, A. O., J. S. Siino, A. A. Gineitis, I. A. Zalenskaya, N. V. Tomilin, P. Yau, and E. M. Bradbury. 2002. Human testis/sperm-specific histone H2B (hTSH2B). Molecular cloning and characterization. J Biol Chem 277: 43474-80. Zhang, H., D. N. Roberts, and B. R. Cairns. 2005. Genome-wide dynamics of Htz1, a histone H2A variant that poises repressed/basal promoters for activation through histone loss. Cell 123: 219-31. 110