Transcripción y Procesamiento del RNA
Anuncio
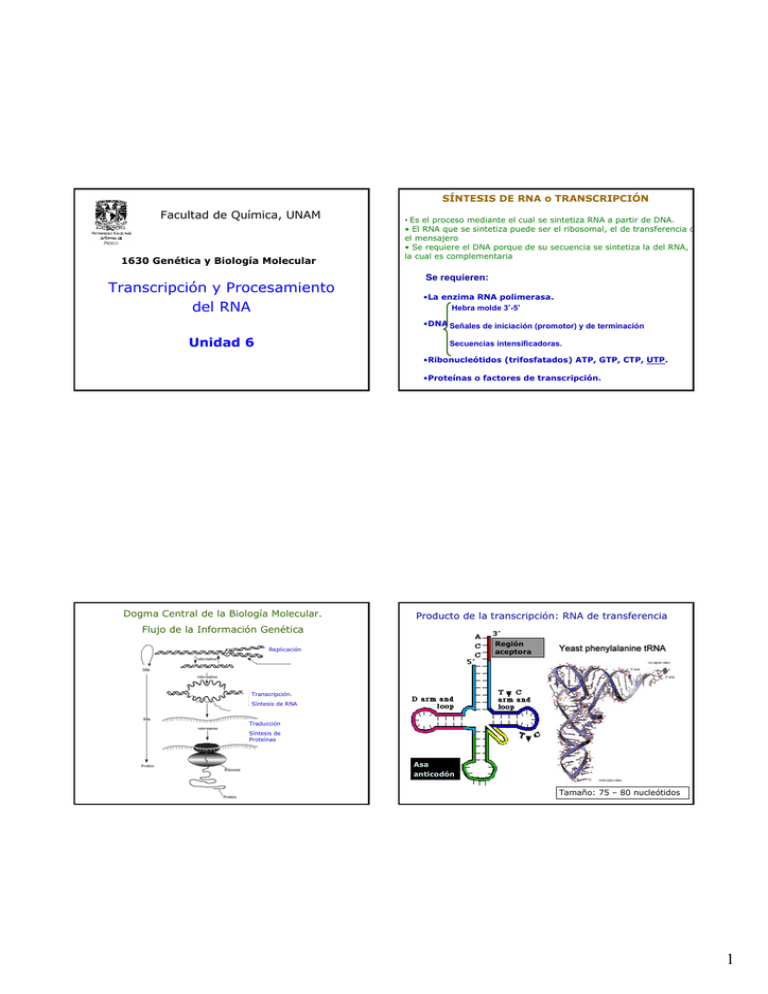
SÍNTESIS DE RNA o TRANSCRIPCIÓN Facultad de Química, UNAM 1630 Genética y Biología Molecular Transcripción y Procesamiento del RNA • Es el proceso mediante el cual se sintetiza RNA a partir de DNA. • El RNA que se sintetiza puede ser el ribosomal, el de transferencia o el mensajero • Se requiere el DNA porque de su secuencia se sintetiza la del RNA, la cual es complementaria Se requieren: •La enzima RNA polimerasa. Hebra molde 3’-5’ •DNA Señales de iniciación (promotor) y de terminación Unidad 6 Secuencias intensificadoras. •Ribonucleótidos (trifosfatados) ATP, GTP, CTP, UTP. •Proteínas o factores de transcripción. Dogma Central de la Biología Molecular. Producto de la transcripción: RNA de transferencia Flujo de la Información Genética Región aceptora Replicación Transcripción. Síntesis de RNA Traducción Síntesis de Proteínas Asa anticodón Tamaño: 75 – 80 nucleótidos 1 Producto de la transcripción: RNA mensajero Estructura y Función del RNAt Los RNAt son las moléculas adaptadoras (traductoras) que decodifican la información en el RNAm acarreando al aminoácido correspondiente. Los RNAt se sintetizan como precursores que son procesados para generar moléculas de 75 a 80 nts de longitud. La estructura de los RNAm es variable y depende de la secuencia. Generalmente tienen bases modificadas como: Ribotimidina Dihidrouridina Pseudouridina Inosina El tamaño de los RNAs mensajeros es variable y depende del tamaño del gen que se transcribe. Inosina Producto de transcripción: El proceso de transcripción es la síntesis de RNA siguiendo un molde de DNA RNA ribosomal PROCARIONTES: RNAr 23S: 2,904 nts. RNAr 16S: 1,542 nts. EUCARIONTES: RNAr 28S: 4,718 nts. Micrografía electrónica de la síntesis de RNA ribosomal RNAr 18S: 1,874 nts. 2 A diferencia de la replicación del DNA, en la transcripción... La enzima que sintetiza RNA es la RNA POLIMERASA La enzima de E. coli está formada por 4 subunidades • Solamente un fragmento de DNA, que corresponde a un gen, es copiado en RNA. • Una de las dos cadenas de DNA es copiada a RNA. DNA Cadena codificante: 5’-ATTCCGATGTACGAGG-3’ DNA 3’-TAAGGCTACATGCTCC-5’ RNA Cadena molde: 5’-AUUCCGAUGUACGAGG-3’ La secuencia de la molécula de RNA que se sintetiza es complementaria y antiparalela a la cadena molde. La molécula de RNA que se sintetiza tiene la misma dirección y secuencia (U -> T) que la cadena codificante. Solamente un fragmento de DNA, que corresponde a un gen, es copiado a RNA. Una de las dos cadenas de DNA es copiada a RNA. Subunidad α: ensamblaje de las unidades y unión al promotor Subunidad β: Sitio catalítico Subunidad β’: Se une al DNA y parte de la subunidad catalítica Subunidad σ: Reconocimiento del promotor específico La subunidad α sirve como nodo para ensamblar la RNA polimerasa holoenzima y esta función reside en el dominio Nterminal de la proteína. • ¿Cómo sabe la RNA polimerasa cuál de las dos cadenas usará como molde? • ¿Cómo sabe la RNA polimerasa dónde comenzar a sintetizar? • ¿Cómo sabe la RNA polimerasa donde terminar de sintetizar? • ¿Quíen abre la doble hélice de DNA? El domino C-terminal de la subunidad α interactúa con la región UP de los promotores que la tengan. 3 La estequiometría de subunidades en la holoenzima es α2ββ'σ Modelo de la RNA polimerasa de E. coli a partir de los datos cristalográficos Núcleo de la enzima Cuando la subunidad σ se asocia al núcleo se forma la holoenzima Núcleo de la enzima α2ββ Estructura de los promotores procariontes. Secuencias que se encuentran “corriente arriba” del sitio de inicio de la transcripción. Hay secuencias muy conservadas en los promotores procariontes. Núcleo RNA pol Holoenzima 5 –8 pb Complejo cerrado Complejo abierto Holoenzima α2ββσ Formación del complejo abierto El factor sigma permite la iniciación de la transcripción permitiendo que la RNA polimerasa se una fuertemente al promotor. Esta unión depende de la apertura de la doble hélice en esa región para formar un complejo de promotor abierto. El factor sigma se disocia de este complejo. • Secuencia -10 o caja Pribnow TATAAT Apertura de la cadena. • Secuencia –35 TTGTCA Es la región de reconocimiento e interacción con el factor σ de la RNA polimerasa. Complejo de elongación. La RNA polimerasa tiene un canal abierto al cual se une el DNA. Una vez que se unen al DNA, los “dedos” de la enzima se cierran alrededor del DNA. 4 Las funciones de la subunidad σ El factor sigma selecciona los genes a transcribirse al facilitar la unión entre la RNA polimerasa y el promotor. Esta unión depende de la denaturalización local del DNA que permite la formación de un complejo de promotor abierto Elongación. RNA polimerasa Rebobinado Desenrollado Hebra codificadora El factor σ se recicla, i.e. cuando se disocia puede ser usado por otra RNA polimerasa. Al unirse al promotor, la RNA polimerasa causa la apertura de al menos 10 - 17 pb de la doble cadena de DNA. Esta “burbuja” de transcripción se mueve con la polimerasa exponiendo la cadena molde, de tal manera que puede ser transcrita. σ ESTRUCTURA DE UNA CADENA DE RNA Hebra molde 3 5 3 5 Hélice híbrida RNA - DNA 5’ ppp RNA naciente 3 Punto de elongación Desplazamiento de la polimerasa 4.- La “burbuja de transcripción” va avanzando, por un lado se abre el DNA duplex y por el otro se re-bobina. 5.- El RNA sintetizado va formando un híbrido (transitorio), con la cadena 3’ – 5’ del DNA BURBUJA DE TRANSCRIPCIÓN Se añaden ribonucleótidos polimerizándose la cadena a través de enlaces fosfodiéster (la cadena va creciendo en la dirección 5’ –> 3’) 5 ELONGACIÓN. 2. Mecanismo independiente de la proteína Rho La subunidad β contiene el sitio activo de la RNA polimerasa donde se forman los enlaces fosfodiéster La secuencia al final del gen contiene repeticiones invertidas que permiten la formación de una estructura de horquilla en el RNA. Hay una región rica en Adeninas, de tal forma que el híbrido DNA-RNA que se forma es débil y se disocia. Hay dos sitios en el DNA que interactúan con la RNA polimerasa: 1. Un sitio de unión débil que involucra la zona del DNA desnaturalizada y el sitio activo en la subunidad β de la polimerasa. La interacciones son principalmente electrostáticas. 2. Un sitio de unión fuerte que involucra al DNA río abajo del sitio activo y lo conforman las subunidades β y β‘ de la enzima. La subunidad β‘ une dos átomos de Zn2+ que participan en la catálisis. Esta subunidad se une fuertemente al DNA. Terminación: Inhibición de la transcripción en procariontes. 1. Mecanismo dependiente de la proteína Rho La rifampicina bloque la transición de iniciación-elongación. Se une a la subunidad β en el complejo RNApolimerasa promotor una vez que se han incorporado dos o tres nucleótidos a la cadena de RNA. La proteína rho es un hexámero que hidroliza ATP en presencia de RNA. Se une al RNA que se está sintetizando y se mueve en dirección al sitio de síntesis. Desestabiliza al híbrido DNA – RNA, facilitando así la terminación de la transcripción. La rifampicina es producida por Streptomyces sp. La estreptolidigina inhibe a la RNA polimerasa durante la elongación. 6 Transcripción en eucariontes. Hay tres actividades de RNA polimerasas RNA polimerasas eucariontes purificadas en una columna de DEAE-Sephadex. RNA pol I, RNA pol II y RNA pol III Las RNA polimerasas eucariontes sintetizan distintos RNAs y difieren en su sensibilidad a inhibidores. INHIBIDORES DE LA TRANSCRIPCIÓN α-amanitina La α−amanitina inhibe a la RNA polimerasa II (Kd =10 nM) y un poco a la III. Inhibe la fase de elongación de la Transcripción Amanita phaloides, hongo venenoso que contiene α - amanitina La RNA polimerasa II eucarionte está formada por 12 subunidades. (P.M. aproximado de 550 kDa) Tres de estas subunidades son homólogas a las subunidades de la RNA polimerasa procarionte. Rpb1 =>β’ Rpb2 =>β Rpb3 =>α Actividad de RNA polimerasa 7 Los promotores de los genes eucariontes son más complejos que los procariontes. Muestran menor conservación en los elementos de reconocimiento de las RNA polimerasas. Inicio de la transcripción 18 - 25 nts Caja TATA: TATA(A/T)A(A/T) Modificaciones posttranscripcionales de RNAs eucariontes *URE (Elementos regulatorios “río arriba”). Son sitios de unión de otras proteínas (factores de transcripción) que facilitan la unión de la RNA polimerasa y la transcripción de ese gen. De 100 a 200 pb del inicio. Enhancers (Sec. Intensificadoras). Regiones en el DNA que están alejadas por más de 1000 pb del sitio de inicio y que activan al promotor para que ocurra una transcripción más eficiente. La caja TATA funciona como señal para la unión de la proteína TBP (TATA-binding protein). La unión de TBP al DNA causa una torsión de éste, facilitando la apertura de la doble hélice. Cada tipo de RNA sufre una forma diferente de PROCESAMIENTO POST-TRANSCRIPCIONAL o MADURACIÓN El factor de transcripción IID (TFIID) se une a promotores que contienen la caja TATA a través de la proteína TBP. RNAt Se forma un complejo multiproteíco en el promotor que permite la asociación de proteínas que están unidas a otras regiones en el promotor. PROCESAMIENTO o MADURACIÓN RNAm Euc. RNAr Remoción de los extremos Modificación de la ribosa Modificación de las bases Empalme o splicing Adición del CAP en el extremo 5’ Adición del poliA en el extremo 3’ Metilación de la ribosa Remoción de partes intermedias de un pre-RNAr largo que origina varios RNAr más cortos 8 RNA ribosomales. Son sintetizados por la RNA pol I. Los RNA ribosomales 28S, 18S, y 5.8S se sintetizan como un transcrito largo de 13,000 nucleótidos que es procesado para generar los RNAr maduros. PreRNA 45S 18S 5.8S RNAm Eucarionte: Adición del CAP 28S • Antes de 20 nucleotidos de largo Metilación en el 2’-OH de la ribosa • Residuo 7-metil guanosina, añadida por una guanililtransferasa • Puente 5´-5´ trifosfato 18S 5.8S corte 28S RNAr maduros RNAr18S RNAr 5.8S Los RNA de transferencia también son procesados • El “capuchón” o “capping” puede estar metilada en O(2´) (1 o dos nucleósidos terminales) o no (cap0). RNAr 28S RNAm eucarionte: Adición de la cola de poliA La RNAsa P hace un corte y genera el extremo 5’-OH Señal de corte Hidrólisis por una endonucleasa específica Secuencias consenso PABP La RNAsa P es un ribozima. Contiena una subunidad de proteína y otra de RNA. Adición de adeninas (50300) por una poli-A polimerasa RNAm poliadenilado 9 RNAm eucarionte: Corte y empalme o splicing RNAm eucarionte: Corte y empalme/ “splicing” DNA Transcripción RNAm heteronuclear Procesamiento: eliminación de intrones. Empalme de exones RNAm maduro RNAm eucarionte: Corte y empalme o splicing RNAm eucarionte: Corte y empalme o splicing Las dos últimas bases del intron son AG Sitio de ramificación Las dos primeras bases del intron son GU 10 El “splicing” del RNAm es catalizado por snRNP (small nuclear RiboNuclear Particles) Comparación de la expresión génica entre eucariontes y procariontes snRNP: Moléculas de RNA ricas en uracilo asociadas a proteínas. U1, U2, U4, U5, U6. La subunidad de RNA forma puentes de hidrógeno con las bases en los sitios de reconocimiento de los intrones (3’, 5’ y la rama). “Spliceosome” = espliceosoma RNA autocatalítico. Algunos intrones se pueden eliminar de transcritos heteronucleares sin el requerimiento de proteínas. En este mecanismo hay un ataque en el sitio 5’ de splicing y la ruptura, y no se forma el lazo (lariat). “Splicing” alternativo. Los RNAhn de algunos genes pueden ser procesados (splicing) de manera diferencial generando dos RNAm maduros distintos. Splicing alternativo Traducción Proteína A Proteína B Por lo general, el resultado del “splicing” alternativo son proteínas relacionadas estructuralmente. Generalmente se obtienen isoformas de una proteína. A partir de un gen, se produce más de una proteína. 11




