Teórica de apoptosis.pdf
Anuncio
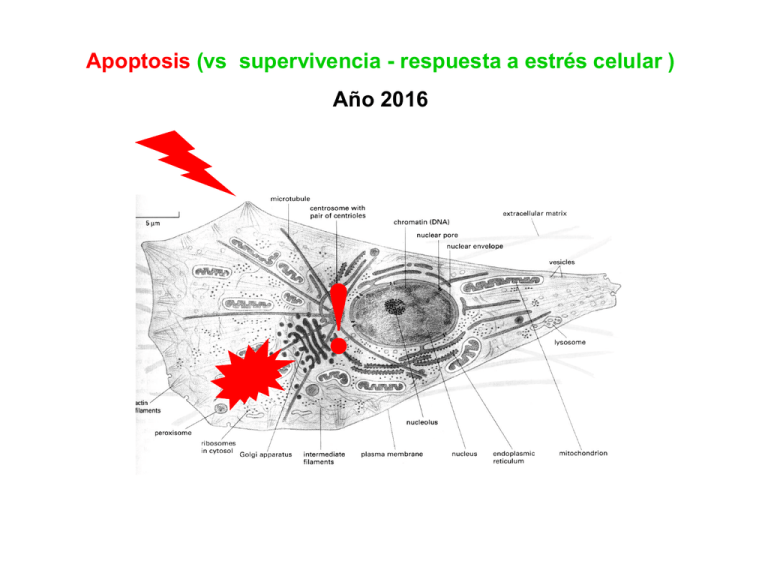
Apoptosis (vs supervivencia - respuesta a estrés celular ) Año 2016 ! Apoptosis: muerte celular programada -en 1972 Kerr et al. describen un tipo de muerte celular distinto de necrosis: apoptosis - Hipócrates de Cos (460-370 AC) describe las gangrenas asociadas a dislocaciones. a p o p t w s i s : “desprendimiento de los músculos de los huesos” Cuando es necesario que las células decidan “suicidarse”? -en adultos, para mantener tamaño de los tejidos -durante el desarrollo, para permitir cambios morfológicos Daño en el DNA alteraciones en ciclo celular estrés celular letal (fuerza o duración) NECROSIS vs APOPTOSIS respuesta celular a una agresión aguda: diversidad de estímulos: -la célula se “hincha y explota” -colapsa el citoesqueleto -inflamación local daño a otras células la célula se encoge y condensa -se disuelve la envoltura nuclear -se fragmenta el DNA -la membrana celular cambia indicando a las células vecinas que puede ser fagocitada -no hay daño a células vecinas -reciclado de componentes Las moléculas efectoras de la apoptosis son las CASPASAS Cisteína en el sitio activo ASPártico es el sitio que atacan con su actividad de proteASA Basal: procaspasas (cerca de 20) inactivas Apoptosis: i) las caspasas efectoras (3, 6 , 7) son activadas por proteólisis mediada por caspasas iniciadoras (2, 4, 8, 9, 10, 12) Cómo se activan las caspasas? 1. mecanismo de activación extrínseco (receptores de membrana) 2. mecanismo de activación intrínseco (mitocondria) puede actuar en forma sinérgica con (1) Familia Bcl-2 (B-cell linfoma-2) (25 genes): -Bcl-2 homology (BH) domains (BH1, BH2, BH3 and BH4), - dominos transmembrana (MOM, ER). - Forman homo-heterodímeros/oligómeros y se activan/inhiben mutuamente Mecanismo de Activación Intrínseco Se activa por daño o disfunciones celulares Ejemplos? Apoptosis disparada por daño al DNA UV Kinasas “sensoras” (ATM ; ATR) Kinasas “efectoras” (CHK1; CHK2) p53 Factor transcripcional genes pro-apoptóticos de la familia Bax Apoptosis y arresto de ciclo celular por daño al DNA UV Kinasas “sensoras” (ATM ; ATR) Kinasas “efectoras” (CHK1; CHK2) p53 1) p21: genes de arresto ciclo celular Factor transcripcional 2) genes pro-apoptóticos de la familia Bax Apoptosis y arresto de ciclo celular por daño al DNA UV p53 Arresto ciclo celular CANCER Apoptosis mecanismo de activación intrínseco (mitocondria) Paso 1: liberación de citocromo C Proteínas de la familia de BCL-2 Bax (asociada a organelas), Bak (interior mit): traslocación a la MOM: poros!!! BCL2: protege membrana mitocondrial Citocromo C Apoptotic Paso 2: activación de caspasa iniciadora 9 Protease Activating APOPTOSOMA Factor (APAF-1) Paso 3: amplificación APOPTOSIS 1. mecanimo de activación extrínseco Ligandos específicos (solubles o no) se unen a receptores de membrana de la familia del receptor de TNF (superfamilia de receptores “death”, 50 miembros) Ejemplos: FAS (CD95) y FASL Trail-R y Trail; TNFR1 y TNF Activación de Receptor DEATH por ligando específico Receptor oligomeriza y recluta proteínas adaptadoras FADD (Fas Associated Death Domain) se une al domino death del receptor Unión de Receptor DEATH por ligando específico Receptor oligomeriza y recluta proteínas adaptadoras DISC: Death Inducing Signaling Complex Procaspasa 8 (iniciadora) se encuentra inactiva Unión de Receptor DEATH por ligando específico Receptor oligomeriza y recluta proteínas adaptadoras DISC: Death Inducing Signaling Complex FADD une moléculas de procaspasa 8, agregándolas. Unión de Receptor DEATH por ligando específico Receptor oligomeriza y recluta proteínas adaptadoras DISC: Death Inducing Signaling Complex La moléculas de procaspasa 8 se activan por proteólisis Unión de Receptor DEATH por ligando específico Receptor oligomeriza y recluta proteínas adaptadoras DISC: Death Inducing Signaling Complex Caspasa 8 activada actúa sobre pro-caspasa 3 (efectora) Unión de Receptor DEATH por ligando específico Receptor oligomeriza y recluta proteínas adaptadoras DISC: Death Inducing Signaling Complex La Caspasa 8 activada cliva y activa caspasa 3 Amplificación Unión de Receptor DEATH por ligando específico (TNF, FasL, etc) Receptor oligomeriza y recluta proteínas adaptadoras DISC: Death Inducing Signaling Complex Se dispara la etapa efectora de la apoptosis: Ej: Caspasa 3 actúa sobre iCAD (Inhidor de Caspase- Activated DNase) Hay interacción entre la vía intrínseca y la extrínseca ? Fas + FASL Proscaspasa 8 caspasa 8 APOPTOSIS (se inserta en MOM) Bid tBid (clivage y reclutamiento en MOM) Bax Bax oligomérico liberación de Citocromo C activación caspasa 9 APOPTOSIS Cómo mantener “silenciados” los factores apoptóticos? Pro-Caspasas: AIPs (Familia Apoptosis Inhibitor Protein) Bad, Bax, etc: miembros anti-apoptóticos de la familia bcl-2 XIAP y IAP/AIP (inhibitor of apoptosis) (familia de prot, pueden ser inducidas por virus) -unen procaspasas e impiden su activación -unen caspasas e impiden su actividad -HSPs: muchas de las proteínas inducidas por estrés son antiapoptóticas. -La vía intrínseca puede ser activada o silenciada por estímulos externos (varias vías de señalización) -la mitocondria y el ER sensan daño y señalizan apoptosis -hay señalización cruzada entre las vías externa e interna Qué cambios se observan en una célula apoptótica? La amplificación de la respuesta lleva a altos niveles de caspasas efectoras. En la fase efectora, la apoptosis es irreversible y se observan cambios morfológicos y moleculares RUPTURA del ADN Caspasa 3 cliva ICAD, Se dispara la fragmentación del ADN Lesiones en ADN activan PARP: la (auto)poly-ADP ribosilación de PARP (múltiples cadenas de polyADPribosa) alertan la maquinaria de reparación de ADN Caspasas clivan PARP e impiden la reparación Cómo visualizar ruptura del ADN? “escaleras” de escalones de 200 nt, tamaño del nucleosoma TUNEL: Terminal deoxynucleotidyl transferase dUTP Nick End Labeling Apoptosis durante la metamorfosis en insectos La membrana plasmática es asimétrica intravesicular / espacio extracelular citosol -glicolípidos -fosfatidilcolina -esfingomielina -fosfatidilserina (carga -) -fosfatidiletanolamina difusión lateral cambio de lado “flip-flop” fexión rotación difusión lateral X cambio de lado “flip-flop” fexión rotación La membrana plasmática es asimétrica espacio extracelular -glicolípidos -fosfatidilcolina -esfingomielina -fosfatidilserina (carga -) -fosfatidilserina (carga -) -fosfatidiletanolamina citosol Apoptosis: fosfatidilserina se expone en el lado extracelular (activación transportador) MW (kD) 10 Molécula FUNCION ??? Hsp10 Constitutivas, abundantes Hsp27 mantiene entramado de microfilamentos de actina Alfa-beta crystallin interfiere activación de caspasa 3. Secuestra factores apoptóticos Bax and Bcl 20-30 10 especies en mamif, ej Hsp27 40 Hsp40 60 Hsp60 (Importación a mitocondrias y cloroplastos) Mantiene integridad de membranan mitocondrial. También es proapoptótica! (se une y contribuye a activar pro-caspasa3) 70 Hsc70, Hsp71, Hsp72, Grp78, BiP Plegado de proteínas. Importación a mitoc y cloroplastos Siempre es inducida por estrés. Fiebre. Manipuleo de muestras in vivo!!! Antiapoptótica: Activa NFKb (anti apoptótico), inhibe JNK (SAPKS) Impide traslocación de Bax a la mitocondria, libetración de citocromo c, formación del apoptosoma, acticavión de pro-caspasas iniciadoras. Mantiene integridad de membrana lisosolam:impide liberación catepsina (………..también tiene rol pro-apoptótico) 90 Hsp90, Grp94 La mas abundante en condiciones basales. (receptores esteroides y factores de transcripción) Antiapoptótica: varios pasos en la activación de NFKB (activación de IKK=inhibición de IKB) 100 Hsp104, Hsp110 Tolerancia a temperaturas extremas Balance entre dos respuestas antagónicas: Stress response Apoptosis -HSPs: hsp 70 inhibe APAF, caspasas, AIF, etc, etc. (Algunas HSPs tienen rol pro-apoptótico) -Regulación traduccional: IRES y uORF traducen genes anti y pro-apoptóticos Balance entre dos respuestas antagónicas: Supervivencia Muerte Bajos niveles de caspasa 3/7 pueden NO terminar en apoptosis supervivencia vs apoptosis durante ER-Stress (Peter Walter) Activación de PERK, IRE 1 ATF4 (transcription factor) IRE1: endonucleasa que inicia el splicing citoplasmático de XBP1/HAC1. IRE1 media la degradación vía RIDD (regulated Ire1dependent decay): se degradan transcriptos traducidos en el ER. Activación transcripcional de BIM (BCL2 pro-apop) XBP1 /HAC1: transcription factor : el mRNA “no-spliceado” está en el citosol La actividad endonucleasa de IRE1 se activa (al igual que en PERK) por liberación de BIP y autofosforilación en trans. Formación de Ire-1 foci (seminario!!!) Activación de IRE1, PERK y ATF6 ATF6: tercer rama de la respuesta a proteínas mal plegadas. ATF4 (transcription factor) Se activa cuando BIP la libera (llega hasta Golgi y se cliva liberando p50 al citosol) p50 es un factor transcripcional P50 y XBP1/HAC1 controlan + y - varios genes IRE-1 y PERK: como contribuyen a la supervivencia y a la apoptosis disparadas por estrés de retículo PERK quimera activable por AP20187 (mediante dimerización de dominios KFBP) (P. Walter Plos One 2009) IRE-1 activable por 1NM-PP1 tg, thapsigargin: inhibe CERCA (bomba de Calcio del ER), desregulando Ca en el ER y afectando chaperonas (Calnexina, calreticulina). Esto induce estrés de retículo. (P. Walter Plos One 2009) Activación prolongada de PERK induce muerte celular (P. Walter Plos One 2009) Activación prolongada de PERK induce apoptosis PERK permite traducción de ATF4, factor de transcripción de CHOP: CHOP es un factor transcripcional que permite la expresión de BIM (miembro pro-apop de la familia de BCL2) (transcripto y proteína inestable) TRAF2 TRAF2: requerido para la activación del receptor de TNF alfa, (vía extrínseca) TRAF2 -TRAF2 también participa en la apoptosis disparada por estrés de retículo Se asocia al ER vía el dominio citosólico de IRE1 (sensa ER-stress): -une y oligomeriza caspasa 12 (se cliva y activa) UPR apoptosis Balance entre dos respuestas antagónicas: Stress response Apoptosis -HSPs: hsp 70 inhibe APAF, caspasas, AIF, etc, etc. (Algunas HSPs tienen rol pro-apoptótico) -Regulación traduccional: IRES y uORF traducen genes anti y pro-apoptóticos -SGs: secuestro de factores pro-apoptóticos Proteínas pro-apoptóticas que van a SGs: TRAF2, otras Críticas a este tipo de experimentos ???????? Ar HS FCCP normal Infección viral respuesta antiviral (interferón, etc) Los SGs son antivirales (varios virus inhiben su formación) Los avSGs son una plataforma para la respuesta antiviral Yoo JS, Takahasi K, Ng CS, Ouda R, Onomoto K, et al. (2014) DHX36 Enhances RIG-I Signaling by Facilitating PKR-Mediated Antiviral Stress Granule Formation. PLoS Pathog 10(3): e1004012. doi:10.1371/journal.ppat.1004012 http://journals.plos.org/plospathogens/article?id=info:doi/10.1371/journal.ppat.1004012 Cuerpos de silenciamiento: ~ “gotas líquidas” que condensan en el citosol “Getting RNA and Protein in Phase” Weber & Brangwynne, Cell 2012 Los SGs están relacionados con agregados de proteínas anómalas involucrados en neurodegeneración Mutantes patogénicas de TDP43 or FUS/TLS inducen SGs TIAR TDP43 Benseñor et al., Curr Chem Biol 2011 Thomas et al, Cell Signal 2011 Diferencias y similitudes entre SGs y agregados proteicos SGs •Inducidos por diversos tipos de estrés (ISR) •Inducidos por inhibición de proteosoma o autofagia •Agregación de PRDs modulada por HSP70 •Proteínas ubiquitinadas •O-glicosilación •Requieren dineina •Altamente dinámicos •Reversibles •acompañan la normal ISR, son protectivos Agregados proteicos / agresomas •Presentes en diversas patologìas (ER-stress y/o OE) •Su disolución requiere degradación por proteosoma o autogafia •Agregación proteínas “mal plegadas” modulada por chaperonas •Contienen ubiquitina •O-glicosilación (ej. Tau) •Requieren dineina •Son muy poco dinámicos •Irreversibles •Presentes en condiciones patológicas (protectivos o patogénicos?) Se investiga actualmente la relevancia funcional (mecanismos de protección celular) y la dinámica de formación y disolución de SGs RNAi screen: 37 novel regulators of SG dynamics in Drosophila S2R+ cells Z-Score > 2 Our screen mammalian 130 fly 2 33 2 3 Ohn et al., Nat Cell Biol 2008 Score = SGs per celldsRNA / SGs per cellcontrol Screens independientes identificaron nuevos pathways Wippich et al., Cell 2013 yeast 101 Buchan et al., Cell 2013
